2020-03-19 東京大学
豊島 有(生物科学専攻 助教)
Wu Stephen (情報・システム研究機構統計数理研究所 データ科学研究系 助教)
吉田 亮(情報・システム研究機構 統計数理研究所 教授)
飯野 雄一(生物科学専攻 教授)
発表のポイント
- 線虫の神経細胞の配置を調べて、個体ごとに配置が大きくばらつくことを見出した。特定の神経細胞に蛍光タンパク質(注1)を発現させて目印とし、配置がばらついていても各々の神経細胞を同定できる方法を開発した。また自動的に神経細胞を同定するソフトウェアも開発した。
- 今回開発した方法によって、ひとつの個体内で同定できる神経細胞の数が大幅に増えた。また線虫の頭部全神経の活動を同時に計測できる技術と統合し、観察した神経活動を回路に対応付けできるようになった。
- 今回の研究によって、全神経活動を同時に計測した結果を神経回路(注2)に対応付け、個体間で比較できるようにする基盤的技術が得られた。神経回路を包括的に観測して動作原理を明らかにすることなどに役立つと期待される。
発表概要
神経細胞に名前をつけて特徴を記載することは、別個体で同じ細胞を特定し(以下同定と呼ぶ)、神経活動などを比較する上で必須である。線虫C.エレガンス(注3)(以下線虫と呼ぶ)では、電子顕微鏡を用いた過去の研究によって、すべての神経細胞に個別に名前がつけられ、細胞の典型的な配置などの特徴が記載された。しかしこうした記載は生きた任意の線虫個体で神経細胞を同定するには不十分であり、一匹の成虫のなかで同定できる細胞の数は限られていた。
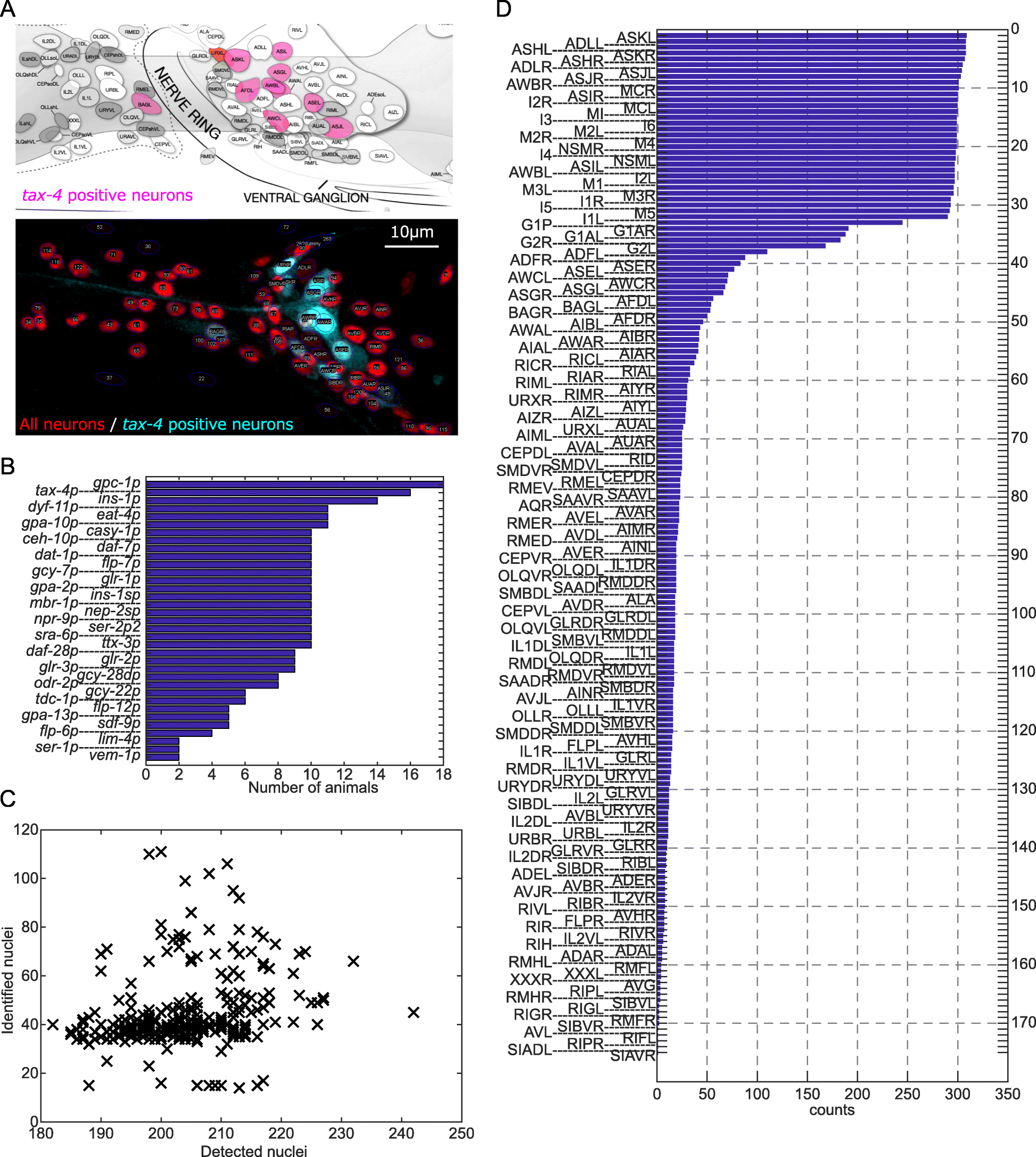
今回、東京大学大学院理学系研究科の豊島有助教らは、特定のパターンで蛍光タンパク質を発現する35種類の線虫株を作出し、311匹の線虫の頭部を詳細に観察した。蛍光タンパク質を発現した細胞をランドマークとして、頭部のほぼ全ての神経細胞を網羅的に同定した。隣接した細胞の距離は平均3.2 μmだったが、各細胞の位置は平均2.7 μmほどばらついていることを見出した(図1)。
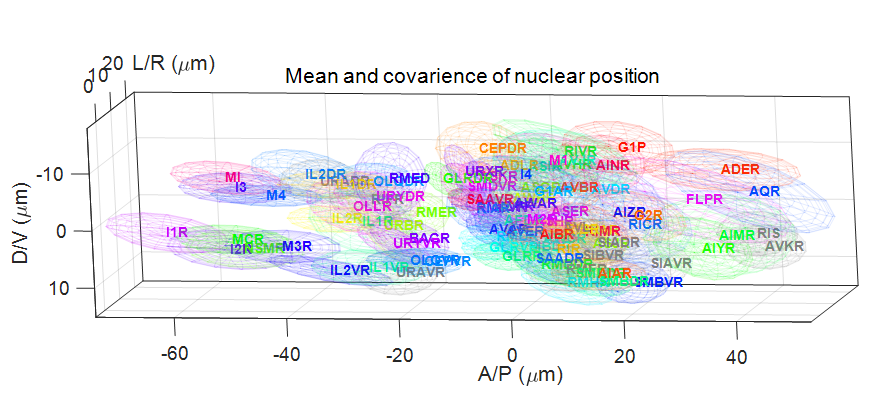
図1:細胞配置のばらつき。各楕円はそれぞれの細胞の核が存在する範囲を示す。見やすさのため、頭部の右半身にある細胞だけを表示した。
こうした個体間のばらつきのため、細胞の配置だけを頼りに細胞を同定することは難しいことがわかった。そこで発現パターンの組み合わせを探索して、細胞同定に最適な線虫株を作出した(図2)。
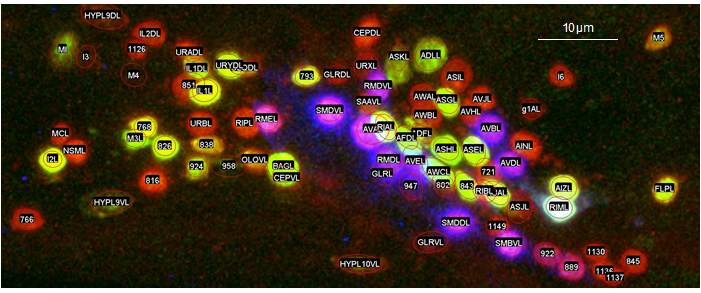
図2:細胞同定に適した線虫株(JN3039)の蛍光パターンの共焦点顕微鏡像。赤や黄色、紫の粒が神経細胞の核で、楕円はその核が検出されたことを示す。数字以外のラベルはその細胞が同定されたことを示す。隣りあった細胞核が塗り分けられ、細胞を同定しやすくなっていることがわかる。見やすさのため、頭部の左半身の最大輝度投影像を表示した。
この線虫株ではひとつの個体内で同定できる細胞の数が従来の3.6倍に増え、線虫頭部のほとんどすべての神経細胞に名前がつけられるようになった。さらに全神経活動の観察ができる線虫株とかけあわせた株でも同様に細胞が同定できることを確かめ、全脳神経活動の同時観測と細胞同定とを統合することに成功した。また豊島助教らは情報・システム研究機構 統計数理研究所のWu Stephen助教らとともに、神経細胞を自動的に同定するソフトウェアを作成した。観察者による修正をフィードバックすることで、正しい細胞同定結果を半自動的に得ることもできた。今回の研究によって、全神経活動を同時に計測した結果を神経回路に対応付けて、個体間で比較するための基盤的技術が得られた。本研究の成果は包括的な観察を通じた神経回路の動作原理の解明などに役立つと期待される。
発表内容
研究の背景
脳・神経系の働きは動物の行動から人間の思考まで多くの生命現象にかかわっており、その動作原理の解明は神経科学の究極の目標の一つである。脳・神経系は複数の神経細胞が互いに配線された回路であり、多様な神経細胞の特徴を調べて同定・分類することで、回路の概要が明らかにされてきた。また例えばある個体内で特徴的な神経活動を示す細胞を見つけたとき、別の個体でも同じ細胞を同定できれば、その特徴的な神経活動が個体に依らない普遍的なものであるかどうかを判断することができる。このように神経細胞の同定は神経科学の研究において必須の過程である。
1980年代の先駆的な研究により、線虫C.エレガンスは118種302個の神経細胞からなるシンプルで個体差の少ない神経回路をもつことが示された。この研究では3匹の成虫を輪切りにして電子顕微鏡で観察し、数年かけて立体再構成することで、神経細胞の典型的な配置や形状、細胞同士の接続などをアトラス(注4)として記載した。ある遺伝子の塩基配列に蛍光タンパク質をつなげて、発現が見られた神経細胞の配置や形状をアトラスと比較することで、その遺伝子を発現する細胞を同定できる。このように特定の細胞で発現を誘導する塩基配列は細胞特異的プロモーターと呼ばれ、蛍光タンパク質の代わりに遺伝子コード型カルシウムセンサー(注5)をつなげば、発現細胞の神経活動を測定することもできる。
一方、近年の顕微鏡技術の発展により、一個体の脳全体を一細胞レベルで可視化する全脳イメージングが可能になった。線虫は体長約1 mmと小さく、体が透明なので、頭部の全神経活動を一細胞レベルで観察することができる。しかし全ての神経細胞が光ってしまうと個々の細胞の形状が得られないなど、全脳イメージングを行った個体内の個々の神経細胞を同定する手法がなく、個体間で神経活動を直接比較することは難しかった。
研究内容
今回、東京大学大学院理学系研究科の豊島 有助教らは、既知の35種類の細胞特異的プロモーターのいずれか1種類で蛍光タンパク質を発現させた一連の線虫株を作出し、計311匹の線虫の頭部を共焦点顕微鏡によって立体的に観察した。線虫頭部には神経細胞やグリア細胞など196個の細胞が密集しているが、これらを見落としなく検出できるソフトウェア RoiEdit3D (Toyoshima et al., 2016)を活用して、観察できた全ての細胞の核の位置と、蛍光タンパク質の発現パターンを調べた。蛍光タンパク質を発現した細胞を目印として、一個体の中で平均44.2個の細胞を同定した。311匹分のデータを合わせると、頭部のほぼ全ての神経細胞を網羅していた。これらをまとめてNeuron IDデータセットとした。
このNeuron IDデータセットを解析したところ、隣接した細胞の核間距離は平均3.2μmだったのに対し、各細胞の核の位置は平均2.7μmほどばらついていた(図1)。つまりある細胞が隣の細胞の位置までずれるようなケースがしばしば生じていることがわかった。このように細胞の位置は個体ごとに大きくばらつくため、位置だけを頼りに細胞を同定することは難しいことがわかった。一方で、咽頭細胞(注6) や側方神経細胞(注7) については相対的な位置関係が保存されており、位置関係に基づいて細胞を同定できる可能性があることもわかった。
つぎに、複数種類の蛍光タンパク質を別々のパターンで発現させて、神経細胞群を塗り分けることで、細胞同定の目印とすることを考えた。蛍光タンパク質が発現していない細胞であっても、周囲の細胞が同定されていれば穴埋め式に同定できる可能性がある。そこで、Neuron IDデータセットを活用して、多数の細胞特異的プロモーターの発現パターンの組合せから、細胞同定に適した組合せを選び出し、実際に線虫に発現させた(図2)。この線虫株(JN3039)では、一個体の中で平均156個の細胞を同定できることがわかった。同定できる細胞数は1種類のプロモーターを用いた場合の3.6倍に増えた。また線虫頭部の196個の細胞のうち186個を同定することができた。さらに、全神経細胞でカルシウムセンサーを発現する線虫株とJN3039株を掛け合わせて、細胞同定付き全脳イメージングに適した線虫株(JN3038)を作出した。全脳イメージングに用いたJN3038株の個体でもJN3039株と遜色ない数の細胞が同定できることを確かめた。
また豊島助教らは、情報・システム研究機構 統計数理研究所のWu Stephen助教らとともに、細胞を自動的に同定するアルゴリズム(注8)を開発した。このアルゴリズムは、Neuron IDデータセットに基づき、細胞の配置のばらつきや相対的位置関係、蛍光タンパク質の発現パターンなどを考慮して、各細胞について同定候補を複数提示することができる。このアルゴリズムを前述のRoiEdit3Dソフトウェアに統合し、細胞核の検出から細胞同定までを一貫して実行できるようにした。自動同定の正答率は61.5%であり、人手による修正の余地があった。RoiEdit3Dでは観察者が自動同定の結果を修正でき、こうした修正を反映して自動同定を繰り返すことで、正しい同定結果を得ることができた。
社会的意義・今後の予定
今回の研究によって、全神経細胞の活動を同時に計測しつつ、同じ個体で大半の神経細胞を同定することで、神経活動を神経回路に対応付けできるようになった。この細胞同定付き全脳イメージング技術を利用すれば、神経活動が回路内を伝わる様子を網羅的に調べることができる。したがって本研究の成果は、こうした包括的な観察を通じた神経回路の動作原理の解明に役立つことが期待される。
発表雑誌
- 雑誌名
BMC Biology論文タイトル
Neuron ID dataset facilitates neuronal annotation for whole-brain activity imaging of C. elegans著者
Yu Toyoshima+, Stephen Wu+, Manami Kanamori, Hirofumi Sato, Moon Sun Jang, Suzu Oe, Yuko Murakami, Takayuki Teramoto, Chanhyun Park, Yuishi Iwasaki, Takeshi Ishihara, Ryo Yoshida*, Yuichi Iino*
※ +: co-first authors, *: co-corresponding authorsDOI番号
10.1186/s12915-020-0745-2
用語解説
注1 蛍光タンパク質
特定の波長の光を当てると別の特定波長の光を発するタンパク質のこと。線虫に組み込んで特定の細胞で発現させることで、その細胞だけを可視化することができる。GFPなどが有名。
注2 神経回路
脳は多くの神経がシナプスによって網の目のようにつながったネットワークを形成している。これが神経回路と呼ばれ、脳が働く基盤となっている。回路を構成する全ての神経細胞の活動を同時に観察することで、回路上の情報の流れなどが精細にわかると考えられている。
注3 C. エレガンス
正式な学名は「Caenorhabditis elegans(カエノラブダイティス・エレガンス)」。小さな線形動物で、バクテリアをエサとする。寿命が短いことや小さな神経系を持つことの利点から、発生生物学や神経科学において広く用いられるモデル生物の一つである。
注4 アトラス
脳など複雑な構造について解剖学的な特徴を包括的に記載した地図のこと。本研究では線虫頭部のすべての神経細胞の位置情報を含んだリファレンスデータを指す。
注5 遺伝子コード型カルシウムセンサー
カルシウムが結合すると蛍光強度が変化するタンパク質のこと。線虫に組み込んで特定の細胞で発現させることで、その細胞の神経活動を測定することができる。
注6 咽頭細胞
咽頭は線虫では口から取り込んだ餌(大腸菌)をすり潰して腸に送る働きをもつ。咽頭内の神経細胞は主に咽頭の動作を制御していると考えられている。
注7 側方神経細胞
線虫頭部の左側または右側に存在する神経細胞群のこと。細胞体は側方神経節とよばれる袋状の構造の内部に存在し、頭部の各所へ神経突起を伸ばしている。
注8 アルゴリズム
特定の目的を達成するための一連の具体的手順のこと。
―東京大学大学院理学系研究科・理学部 広報室―