2020-03-03 筑波大学,理化学研究所
研究成果のポイント
1. 単一細胞の RNA 解析においてゲノム上のどの場所から RNA が出ているのかを示すリードカバレッジを可視化するソフトウェア「Millefy」を新規開発し、ES 細胞やがん細胞で、リードカバレッジに細胞ごとのばらつきが見られることを明らかにしました。
2. リードカバレッジは、RNA の維持などの生命現象を反映する重要な情報です。本研究により、ES 細胞やがん細胞での様々な現象を発見できるようになりました。
3. 発生・再生研究や疾患研究において、新たなバイオマーカーや RNA 関連現象、疾患関連現象の発見に貢献すると期待されます。
国立大学法人筑波大学 医学医療系・人工知能科学センターの尾崎遼准教授(国立研究開発法人理化学研究所 生命機能科学研究センター 客員主幹研究員)、国立研究開発法人理化学研究所 生命機能科学研究センターの二階堂愛チームリーダーらの研究グループは、1細胞RNAシーケンス法(1細胞 RNA-seq)注1)データのリードカバレッジ注2)の細胞間変動(細胞ごとのばらつき)を可視化するソフトウェア
「Millefy(ミルフィー)」を開発しました。
1細胞RNA-seqデータのリードカバレッジには、既知・新規転写単位、正常・異常スプライシング、アンチセンスRNAやエンハンサーRNAの発現などの様々なRNA関連生命現象が反映されています。これらの生命現象の細胞間変動は、発生・再生過程や疾患において観察される、細胞間での機能の違いに寄与すると考えられるため、リードカバレッジの細胞間変動の可視化は、疾患機序解明やバイオマーカー探索において重要です。しかし、これを効果的に捉えるデータ可視化手法はこれまで存在しませんでした。
本研究では、リードカバレッジの細胞間変動を可視化するソフトウェア「Millefy(ミルフィー)」を開発しました。Millefyを、既報のマウス胚性幹細胞(ES細胞)およびトリプルネガティブ乳がん注3)患者由来上皮細胞の1細胞RNA-seqデータに適用したところ、アンチセンスRNAの制御、エンハンサーRNAの発現、非翻訳領域長といった、遺伝子発現量だけでは発見・解釈が困難な細胞間変動を明らかにできることがわかりました。
本研究の成果は、発生・再生研究、がんなどの疾患研究、神経科学といった、1細胞RNA-seqが盛んに用いられている分野において、バイオマーカーの探索や新規機序の発見に貢献すると期待されます。
本研究の成果は、2020年3月3日付「BMC Genomics」で公開される予定です。
* 本研究の一部は、理化学研究所基礎科学特別研究員制度(尾崎遼)、科学技術振興機構(JST)戦略的創造研究推進事業(CREST)「統合1細胞解析のための革新的技術基盤(研究統括:菅野純夫)」の研究課題「臓器・組織内未知細胞の命運・機能の1細胞オミクス同時計測(研究代表者: 二階堂愛)」、JSTおよび日本医療研究開発機構(AMED)再生医療実現拠点ネットワークプログラム「iPS・分化細胞集団の不均質性を1細胞・全遺伝子解像度で高速に測定する技術の開発(研究代表者: 二階堂愛)」「超多検体オミクスによる細胞特性の計測(研究代表者: 二階堂愛)」の支援を受けて行われました。
研究の背景
1細胞RNAシーケンス法(1細胞RNA-seq)は細胞ごとにRNAの量や種類を計測する技術であり、発生・再生生物学や疾患研究において細胞集団の組成解明、細胞機能の特定、バイオマーカーの探索などに用いられています。
1細胞RNA-seqのデータ解析では、遺伝子発現量行列注4)に対してPCA(主成分分析)などの次元圧縮を適用することで、細胞集団の構造を可視化することが一般的です。
一方で、これらの可視化手法で無視されてきたのが、リードカバレッジです。リードカバレッジは、ゲノム上のどの領域からどのくらいRNAが転写されたかを表すシグナルの分布です。1細胞RNA-seqのリードカバレッジはRNAに関連する様々な生命現象(例えば、既知および新規の転写領域、選択的スプライシング、疾患などにおけるスプライシング異常、イントロンリテンション、アンチセンスRNAやエンハンサーRNAなどの非コードRNAの発現)を反映していると考えられていますが、このような現象は、リードカバレッジの形状などの情報が捨象される遺伝子発現量行列の解析のみでは見逃されるリスクがあります。また、リードカバレッジの可視化は実験技術や情報解析技術の検証にも有用です。
これまでに、1細胞RNA-seqによって、遺伝子発現に細胞ごとのばらつき(細胞間変動)があることやその意義が明らかにされてきました。したがって、1細胞RNA-seqのリードカバレッジを効果的に可視化することができれば、そこに反映される様々な生命現象の細胞間変動の発見・解釈につながると考えられます。その際、(1)多数の細胞のリードカバレッジを一覧できること、(2)ゲノムのコンテキスト(ゲノム上の各領域における遺伝子やエピゲノムなどの注釈)とリードカバレッジを関連づけて表示できること、(3)細胞間変動を見やすく強調できることが、重要な要件となります。しかしながら、これらの要件を満たすデータ可視化手法はこれまで存在しませんでした。
研究内容と成果
本研究グループは、1細胞RNA-seqのリードカバレッジを効果的に可視化するソフトウェア「Millefy(ミルフィー)」を開発しました。Millefyは、各細胞のリードカバレッジをヒートマップ注5)およびゲノムブラウザ注6)のように表示し、さらに局所的なリードカバレッジの細胞間変動に応じて細胞を自動で並べ替える機能を備えています。細胞の自動並べ替えには、拡散マップという教師なし非線形次元圧縮手法を援用しています。これらの機能により、上述の3要件を満たすことに成功しました(参考図1、2)。
Millefyの有効性を示すため、まず、既報のマウス胚性幹細胞(ES細胞)への分化誘導後の時系列1細胞RNAseqデータにMillefyを適用しました。その結果、例えばZmynd8遺伝子の長鎖型アイソフォーム注7)とそのアンチセンスRNAであるZynd8asのリードカバレッジの細胞間変動から、両者が互いに異なる制御を受けることが示されました(参考図3)。このような知見は、細胞のリードカバレッジを時刻ごとに平均するだけでは見分けるのが難しく、本手法の重要性を示唆するものです。
また、Myc遺伝子の周辺にあるエンハンサー領域(遠位の転写活性化領域)を可視化したところ、エンハンサー領域由来のRNA(エンハンサーRNA)が細胞間変動を示していることがわかりました(参考図4)。エンハンサーRNA はエンハンサー活性の指標になったり遺伝子発現制御に関与することが知られているものの、多くは遺伝子データベースに含まれていないために、一般的な1細胞RNA-seqデータ解析では見逃されています。この結果は、1細胞 RNA-seqのリードカバレッジとともに遺伝子およびエンハンサー領域のアノテーションを表示することで、遺伝子領域外におけるRNA転写現象に解釈を与えうることを意味しています。
さらに、既報のトリプルネガティブ乳がん患者由来の上皮細胞の1細胞RNA-seqデータにMillefyを適用しました。 Millefyでc-JUNやNRAS注8)の遺伝子座のリードカバレッジを可視化した結果、3′側の非翻訳領域(3′UTR)注9)の短縮が細胞によってばらつきを持ってみられることがわかりました(参考図5)。これらの遺伝子の3′UTRの短縮は、高い浸潤性との関連が報告されており、今回の結果は腫瘍組織内における腫瘍細胞間の浸潤性のばらつきとの関連している可能性があります。なお、このような3′UTRの長さの細胞間不均一性は、元のデータを報告した論文では指摘されていませんでした。
さらに、異なる実験プロトコルによる1細胞RNA-seqのデータにMillefyを適用することで、長鎖RNAの測定性能がプロトコル間で異なることを明らかにし、リードカバレッジの細胞間変動の可視化が品質管理ツールとしても役立つことがわかりました。
これらの結果から、既存の解析手法では見逃されるようなものであっても、Millfyがリードカバレッジに反映される様々な生命現象の細胞間変動を探索するツールになることを示しています。
今後の展開
Millefy によるリードカバレッジの可視化は、1細胞 RNA-seq 実験技術の開発・導入において、遺伝子発現量行列を対象とした既存のデータ解析パイプラインを補完するツールとして活用できると考えられます(参考文献1)。また、AI などの情報解析技術によって選択的スプライシングや新規RNAを検出するソフトウェアの開発においても、予測結果の検証のためにリードカバレッジの可視化は必要とされます(参考文献2)。
1細胞RNA-seqは、発生・再生研究、がんなどの疾患研究、神経科学など様々な研究分野で、盛んに用いられていており、そのデータは今後も増大することが予想されます。また、がんや発生生物学において、遺伝子発現量の変化だけではなく、スプライシングの制御や破綻、アンチセンス RNA やエンハンサーRNA などの非コード RNA の重要性が明らかになりつつあり、Millefy は、リードカバレッジの細胞間不均一性の可視化を通じて、新規機序の理解やバイオマーカー探索といった形で正常・疾患細胞集団に着目する多様な研究分野に貢献すると期待されます。
Millefy は、R のパッケージや Docker イメージといった、医学生物学分野で親しまれている使いやすい配布形式で提供されており(https://github.com/yuifu/millefy)、アカデミアや企業において1細胞RNA-seqに携わる人がすぐに使うことができます。
参考図
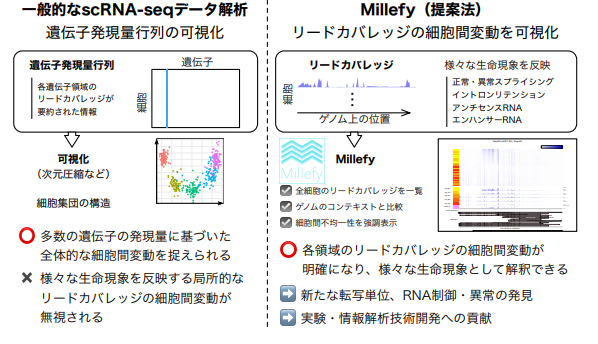
図1 本研究の概要
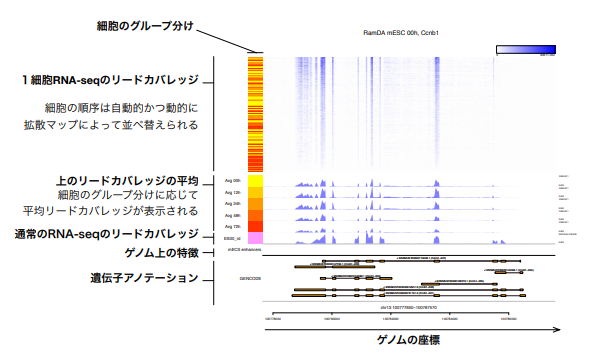
図2 Millefyの概要
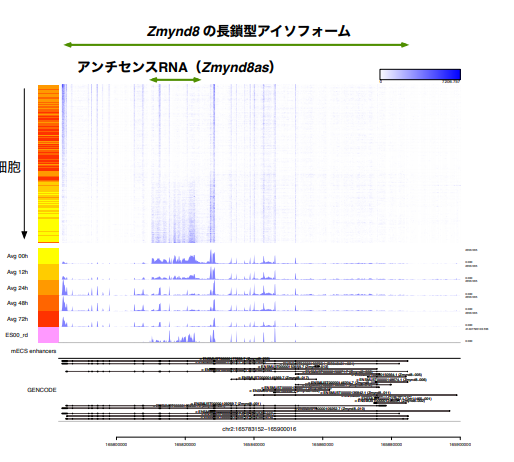
図3 MillefyでZmynd8遺伝子座を可視化した例
マウス胚性幹細胞(ES 細胞)に分化誘導後の時系列1細胞 RNA-seq データに Millefy を適用した。アンチセンスRNAの領域と長鎖アイソフォームの領域が異なる制御を示すことがわかる。
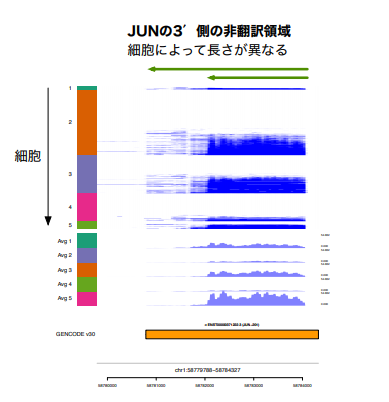
図4 トリプルネガティブ乳がん患者由来の上皮細胞の1細胞 RNA-seq データに Millefy を適用した例細胞によって c-JUN 遺伝子の 3′側の非翻訳領域の長さが異なる様子がわかる。
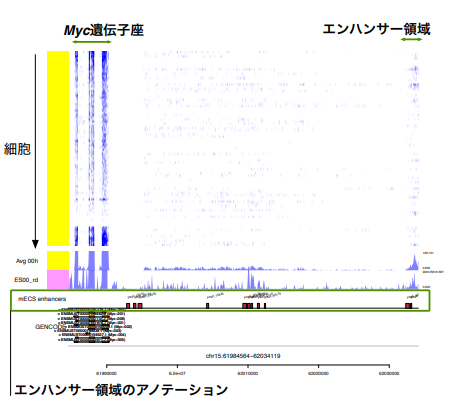
図5 Millefy によるエンハンサー領域の可視化
エンハンサー領域のアノテーションを同時に表示することで、リードカバレッジの中からエンハンサーRNA を見出すことができる。
用語解説
注1) 1細胞 RNA シーケンス法(1細胞RNA-seq)
単一細胞に含まれる RNA の種類と量を網羅的に測定する技術。臓器や腫瘍組織に含まれる細胞集団の組成の理解、生命現象の機序の解明、バイオマーカーの探索などに使われる。近年、発生生物学、再生医学、疾患研究などの分野で広く用いられている。
注2) リードカバレッジ
ゲノム上のどの場所からどのくらいRNAが転写されたかを表すシグナルの分布。1細胞RNA-seq のリードカバレッジは、転写領域、選択的スプライシング(遺伝情報の切断・再結合)、疾患におけるスプライシング異常、アンチセンス RNA(特定の遺伝子発現を阻害する RNA)やエンハンサーRNA(遺伝子発現を調節するゲノム領域の制御に関与する RNA)などの非典型的な RNA 合成、といった様々な生命現象を反映している。
注3) トリプルネガティブ乳がん
乳がん全体の約 10〜20%を占める、乳がんのタイプの一つ。エストロゲンやプロゲステロンにより増殖する性質を持たず、かつ、がん細胞の増殖に関わる糖タンパク HER2 を過剰に持たないため、ホルモン療法や抗 HER2 療法が有効でない。
注4) 遺伝子発現量行列
細胞数×遺伝子数の大きさの行列、各細胞における各遺伝子の発現量を記録したデータ。1細胞 RNA-seq データ解析においてよく用いられる。遺伝子発現量は、各遺伝子領域におけるリードカバレッジのシグナルの(エキソン領域について)和をとったものと解釈でき、リードカバレッジのシグナルの形状を捨象した情報と捉えることができる。
注5) ヒートマップ
表形式のデータをマス目の各目の値に応じて色を変えて表示する可視化手法。
注6) ゲノムブラウザ
ゲノム座標に対し、様々な計測データや注釈データを表示する可視化手法。
注7) 長鎖型アイソフォーム
一種類の遺伝子から複数種類の RNA ができるとき、それぞれの RNA をアイソフォームと呼ぶ。このうち、RNA 分子長が相対的に長いアイソフォームを特に長鎖型アイソフォームと呼ぶ。
注8) c-JUN および NRAS
c-JUN はDNAに結合して遺伝子発現を制御する転写因子である。NRASはシグナル伝達分子で細胞増殖を制御する。いずれのタンパク質をコードする遺伝子も、変異が入るとがん化に寄与するがん関連遺伝子と考えられている。
注9) 3′側の非翻訳領域(3′UTR)
タンパク質をコードする遺伝子由来 RNA の中でタンパク質の情報が含まれていない領域を非翻訳領域(UTR)と呼ぶ。RNA 分子が合成される時に先に合成される側を 5′側、後に合成される側を 3′側と呼ぶことから、3′に位置する非翻訳領域を特に 3′UTR と呼ぶ。
参考文献
1. Hayashi T, Ozaki H, Sasagawa Y, Umeda M, Danno H, Nikaido I. 462 Single-cell full-length total rna sequencing uncovers dynamics of 463 recursive splicing and enhancer rnas. Nat Commun. 2018;9(1):619.
2. Matsumoto H, Hayashi T, Ozaki H, Tsuyuzaki K, Umeda M, Iida T, Nakamura M, Okano H, Nikaido I. A nmfbased approach to discover overlooked differentially expressed gene regions from single-cell rna-seq data.
NAR Genomics Bioinforma. 2020;2(1):020.
掲載論文
【題 名】 Millefy: visualizing cell-to-cell heterogeneity in read coverage of single-cell RNA sequencing datasets
(Millefy による1細胞 RNA-seq データにおけるリードカバレッジの細胞間変動の可視化)
【著者名】 Haruka Ozaki, Tetsutaro Hayashi, Mana Umeda, Itoshi Nikaido
【掲載誌】 BMC Genomics (DOI: 10.1186/s12864-020-6542-z)
問合わせ先
尾崎 遼(おざき はるか)
筑波大学 医学医療系 バイオインフォマティクス研究室 准教授