Split-CRISPR-Cpf1の開発
2019-08-13 東京大学,科学技術振興機構
ポイント
- 新たなゲノム編集ツールとして注目されているCpf1タンパク質を二分割したsplit-Cpf1を新たに開発した。
- Split-Cpf1を用いて、光刺激による精度の高いゲノム編集や、極めて高い効率で遺伝子発現を制御することに成功した。
- Split-Cpf1によるゲノム編集の技術開発により、CRISPR-Cpf1によるゲノムエンジニアリングが今後、さらに展開することが期待される。
東京大学 大学院総合文化研究科の二本垣 裕太 大学院生(現 ジョンズホプキンズ大学 医学部 細胞生物学科 博士研究員)、小田部 尭広 特任研究員、佐藤 守俊 教授らの研究グループは、Cpf1タンパク質を二分割して得た分割体(split-Cpf1)に基づいて、ゲノム(遺伝子)編集を光によって制御したり、より効率的に遺伝子を発現したりできるツールを開発することに成功しました。
近年報告されたCRISPR-Cpf1注1)は、従来技術のCRISPR-Cas9注2)よりも標的DNAへの特異性が高いため、オフターゲット効果注3)が小さく精度の高いゲノムエンジニアリング技術になると期待されています。しかし、Cpf1の分子構造がCas9とは大きく異なるため、Cas9を基に開発されてきた応用技術が、そのままではCpf1に適用できないという問題がありました。本研究では、この問題を解決するためにCpf1を分割してsplit-Cpf1を開発し、光刺激による精度の高いゲノム編集や極めて高い効率での遺伝子発現制御を実現しました。Split-Cpf1を基盤として、生体でのゲノムエンジニアリング注4)がさらに発展することが期待されます。
本研究成果は、米国科学誌「Nature Chemical Biology」(電子版:英国時間8月12日)に掲載されます。
本研究成果は、国立研究開発法人 科学技術振興機構(JST)の戦略的創造研究推進事業(CREST)「光の特性を利用した生命機能の時空間制御技術の開発と応用」(研究総括:影山 龍一郎 京都大学 ウイルス・再生医科学研究所 教授)における「ゲノムの光操作技術の開発と生命現象解明への応用」(研究代表者:佐藤 守俊 教授)および同 大学発新産業創出プログラム(START)注5)「CRISPR-Cas9システムを光制御するゲノムエンジニアリングツール」(研究代表者:佐藤 守俊 教授)の一環として得られました。
<研究背景>
ゲノム(遺伝子)編集技術としてCRISPR-Cas9が発表されて以来、ゲノム上の狙った遺伝子の機能を破壊したり(ノックアウト)、別の塩基配列に置き換えたりすること(ノックイン)が簡単にできるようになりました。また、Cas9を用いた応用技術により、ゲノム上の狙った遺伝子の発現量を上げたり、下げたりすることも可能になりました。このようなCas9に基づくゲノムエンジニアリング技術は、基礎研究から医療や創薬、食物の品種改良といった身近な分野にまで広く応用されています。
近年、CRISPR-Cas9に加えて、CRISPR-Cpf1がゲノム編集技術として利用できることが報告されました(参考文献1、2、3、4)。Cpf1は、ガイドRNAと結合し、ガイドRNAの一部と相補的な二本鎖DNAを認識します。Cpf1は、Cas9と比べて標的DNAへの特異性が高く、オフターゲット効果が小さいことが大きな特徴です。また、ガイドRNAを自身で切り出せる機能を持っていて、複数の遺伝子を同時に標的にすることもできます。しかし、Cpf1の分子構造はCas9とは大きく異なるため、Cas9を基に開発されてきた応用技術が、そのままではCpf1に適用できないという問題がありました。
<研究内容>
本研究グループは、上述の問題を解決するために、Cas9を分割することで化合物または光刺激によりゲノム編集を誘導できることに着目し、まずCpf1をさまざまな箇所で二分割しました。得られた34種類の分割体(split-Cpf1)の中には、そのままではN末端断片とC末端断片注6)が会合しないものが含まれていました。分割したsplit-Cpf1のN末端断片とC末端断片が会合しないと、標的の遺伝子に結合できないことから、会合を外部から誘導することが必要です。この“誘導型のsplit-Cpf1”に、本研究グループが開発した光スイッチタンパク質“Magnetシステム”注7)(参考文献5)を連結することで、青色光のオン/オフによりDNA切断活性をオン/オフに誘導することができる光活性化型のCpf1(paCpf1)を開発しました(図1)。そこで、ヒトDNMT1遺伝子注8)を標的としたゲノム編集を指標としてpaCpf1を評価したところ、暗所ではpaCpf1は全くDNA切断活性を示さずヒトDNMT1遺伝子の塩基配列を編集しませんでしたが、青色光を照射するとDNA切断活性が観察され、当該遺伝子のゲノム編集を実行できることが確認できました。また、paCpf1のオフターゲット効果を検証したところ、paCpf1のオフターゲット効果は、二分割しても、Cpf1と同程度に小さいことも明らかになりました。
今回、Cpf1の二分割で得られたsplit-Cpf1の中には、上述の“誘導型のsplit-Cpf1”に加えて、N末端断片とC末端断片が自然と会合して機能する“自発会合型のsplit-Cpf1”も含まれていることが分かりました。本研究グループは、Cas9について“誘導型”の分割体(split-Cas9)を報告していますが(参考文献6、7)、CRISPRシステムにおいて“自発会合型”として細胞内で高いDNA切断活性を示したのは、本研究の“自発会合型のsplit-Cpf1”が初めてです。
本研究グループでは、“自発会合型のsplit-Cpf1”を用いて、極めて高い効率で遺伝子発現を活性化できる技術を開拓しました。“自発会合型のsplit-Cpf1”には、Cpf1が本来持っているN末端とC末端に加えて、分割によって新しく生じたN末端とC末端があります。Split-Cpf1のDNA切断活性を欠失させる変異を導入した(“split-dCpf1”と呼ぶ)上で、split-dCpf1の4つの末端の全てに転写活性化因子注9)と核局在化シグナル配列注10)を連結し、遺伝子発現の活性化技術として“dCpf1-SA2.0”を開発しました(図2)。培養細胞における内在性遺伝子の発現を指標として当該技術を評価したところ、“dCpf1-SA2.0”は、CRISPR–Cas9を用いた従来技術(参考文献8)よりもはるかに高い効率で遺伝子発現を活性化できることが分かりました。
さらに、本研究グループは、マウスの生体内でも“dCpf1-SA2.0”が効率よく遺伝子発現を活性化できることを示しました。マウスの肝臓に“dCpf1-SA2.0”を導入したところ、当該臓器でルシフェラーゼ注11)のタンパク質が効率よく発現していることが分かりました(図3a、b)。ちなみに、dCpf1を用いた既存技術(dCpf1-VPR)(参考文献9)では、ルシフェラーゼの発現がほぼ観察できませんでした(図3a、b)。また、内在性遺伝子の発現を指標に評価したところ、“dCpf1-SA2.0”は、当該遺伝子の発現をマウスの生体内で非常に強く活性化できることが示されました(図3c)。
上述のように本研究グループは、新たに“誘導型のsplit-Cpf1”による青色光によるゲノム編集の光操作技術(paCpf1)を開発するとともに、“自発会合型のsplit-Cpf1”に基づいて、遺伝子発現の活性化技術(dCpf1-SA2.0)を開発しました。本研究により、光刺激による精度の高いゲノム編集や極めて高い効率での遺伝子発現を制御することができます。今後、split-Cpf1を基盤として、生体でのゲノムエンジニアリングがさらに発展するとともに、生命科学の原理・現象の解明につながることが期待されます。
<参考図>
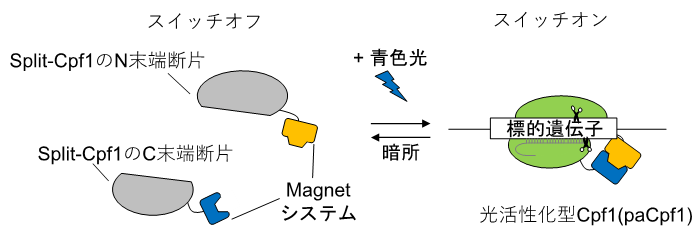
図1 本研究で開発した光活性化型Cpf1(paCpf1)の模式図
Split-Cpf1のN末端断片とC末端断片に光スイッチタンパク質(Magnetシステム)を連結して、光活性化型Cpf1(paCpf1)を開発した。paCpf1は青色の光を照射すると、Magnetシステムの結合に伴って、本来のCpf1のようにDNA切断活性を回復し、標的遺伝子を切断する(スイッチオン)。青色光の照射を止めると、Magnetシステムが乖離し、DNA切断活性を失う(スイッチオフ)。
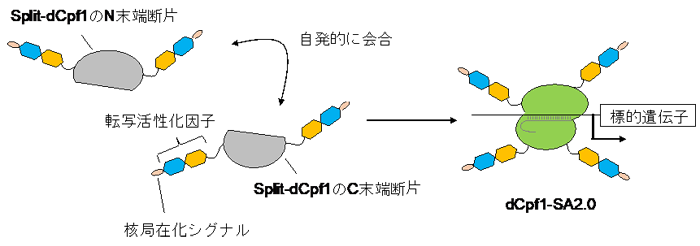
図2 本研究で開発した自発会合型Cpf1(dCpf1-SA2.0)
Split-dCpf1には、本来のN末端とC末端に加えて、分割によって新しく生じたN末端とC末端がある。この4つの末端の全てに転写活性化因子および核局在化シグナル配列を連結することで、標的遺伝子を強力に活性化することが可能になった。
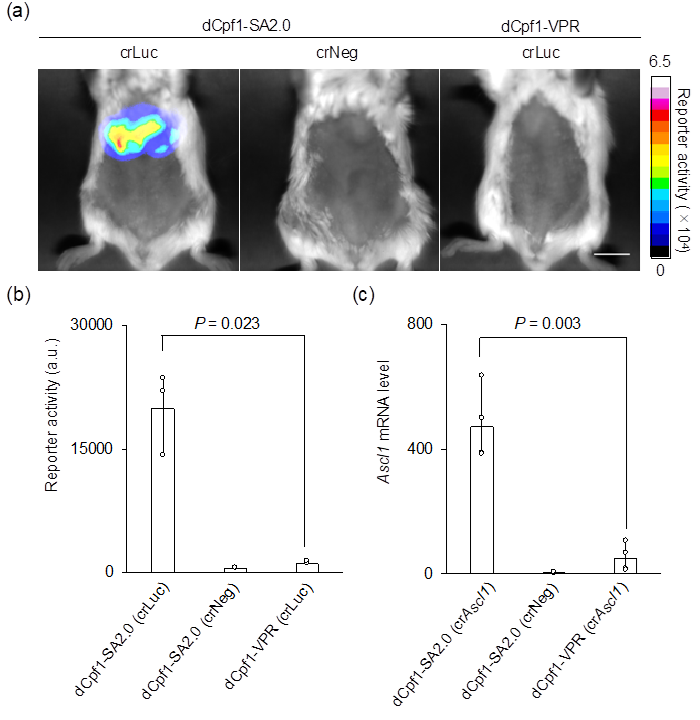
図3 マウスの肝臓におけるルシフェラーゼレポーターの応答
(a)マウスの肝臓での遺伝子発現を、ルシフェラーゼをレポーターとして用いることで可視化した。dCpf1-SA2.0は、ネガティブコントロール(crNeg)およびdCpf1を用いた既存技術(dCpf1-VPR)と比べて、非常に高い効率でルシフェラーゼの発現を誘導した。
(b)図3aの画像データから遺伝子発現を数値データで示している。
(c)内在性遺伝子(Ascl1)の発現を指標に評価した結果、dCpf1-SA2.0は、当該遺伝子の発現を、マウスの肝臓で、既存技術(dCpf1-VPR)よりもはるかに強く、活性化できることが示された。
<用語解説>
- 注1)CRISPR-Cpf1
- ゲノムの切断を人為的に行うための技術。Cpf1と呼ばれるDNA切断酵素がガイドRNAとともにDNAに結合し、そのDNA配列を部位特異的に切断する。このCRISPR-Cpf1システムは、バクテリオファージに対する原核生物の免疫システムとして発見された。CRISPR-Cas9と同様にゲノム編集に利用されている。
- 注2)CRISPR-Cas9
- ゲノムの切断を人為的に行うための技術。Cas9と呼ばれるDNA切断酵素がガイドRNAとともにDNAに結合し、そのDNA配列を部位特異的に切断する。このCRISPR-Cas9システムは、バクテリオファージに対する原核生物の免疫システムとして発見されたが、2012年以降、ゲノム編集に利用されている。
- 注3)オフターゲット効果
- 本来の標的とする配列以外の類似配列を認識してしまい、意図しない変異が生じてしまうこと。
- 注4)ゲノムエンジニアリング
- ゲノム上の遺伝子の塩基配列を改変してその機能を破壊したり(ノックアウト)、別の塩基配列で置き換える(ノックイン)技術や遺伝子発現の亢進・抑制をしたり、特定の遺伝子を書き換える技術のこと。
- 注5)大学発新産業創出プログラム(START:Program for Creating STart-ups from Advanced Research and Technology)
- 日本の大学などの基礎研究成果に関し、大学等発ベンチャーなどを通じた新規マーケットへの事業展開が十分に行われていない現状を踏まえて、平成24年度に文部科学省により「大学発新産業創出拠点プロジェクト」として創設され、平成27年度より科学技術振興機構が実施している制度。本制度では、事業化ノウハウを持った人材(事業プロモーター)ユニットを活用して、大学などのポテンシャルの高いシーズの事業化を通じて新産業の創出、新規マーケットの開拓を目指す。大学等発ベンチャーの起業前段階から公的資金と民間の事業化ノウハウを組み合わせることにより、事業戦略・知財戦略を構築しつつ、既存企業にはリスクの負えないポテンシャルの高いシーズの事業化への挑戦を支援する。本プログラムの成果として「遺伝子や生命の働きをコントローラブルに」をコンセプトとした大学発ベンチャー「株式会社ミーバイオ」が平成31年4月1日に設立された。本ベンチャーは遺伝子改変動物を提供する事業からスタートし、特定疾患モデルマウスの受託開発や受精卵のライセンス販売を目指す。
- 注6)N末端断片とC末端断片
- 酵素(タンパク質)はアミノ酸のアミノ基とカルボキシ基がペプチド結合でつながったポリマー。このポリマーの末端のアミノ基側を含む部分をN末端断片、カルボキシ基側を含む部分をC末端断片と呼ぶ。
- 注7)Magnetシステム
- Magnetシステムは、アカパンカビ(Neurospora crassa)が有する小さな光受容体のヴィヴィッド(Vivid)に対して多角的にプロテインエンジニアリングを施して開発されたタンパク質の対である。暗所では単量体として存在し、青い光を受容するとヘテロ二量体を形成する。
- 光による単量体と二量体の変換を利用して、さまざまな光活性化型のツールを設計・開発することができる。
- 注8)DNMT1遺伝子
- ヒト19番染色体にコードされた遺伝子。DNAのメチル化の制御に関わる。
- 注9)転写活性化因子
- 転写を開始するために必要な複合体を呼び寄せて、転写を活性化するための因子のこと。
- 注10)核局在化シグナル配列
- 細胞質で合成されたタンパク質を核に運び、局在化させるアミノ酸配列のこと。
- 注11)ルシフェラーゼ
- ホタルや発光キノコなどの、生物発光を触媒する酵素のこと。遺伝子発現のレポーターとして使われている。
<論文タイトル>
- “A split CRISPR–Cpf1 platform for inducible genome editing and gene activation”
- 著者名:Yuta Nihongaki, Takahiro Otabe, Yoshibumi Ueda, Moritoshi Sato
- DOI:10.1038/s41589-019-0338-y
<参考文献>
1)Zetsche, B. et al. Cpf1 is a single RNA-guided endonuclease of a class 2 CRISPR-Cas system. Cell, 163, 759–771 (2015).
2)Kleinstiver, B. P. et al. Genome-wide specificities of CRISPR-Cas Cpf1 nucleases in human cells. Nat. Biotechnol., 34, 869–874 (2016).
3)Kim, D. et al. Genome-wide analysis reveals specificities of Cpf1 endonucleases in human cells. Nat. Biotechnol., 34, 863–868 (2016).
4)Yamano, T. et al. Crystal structure of Cpf1 in complex with guide RNA and target DNA. Cell, 165, 949–962 (2016).
5)Kawano, F. et al. Engineered pairs of distinct photoswitches for optogenetic control of cellular proteins. Nat. Commun., 6, 6256 (2015).
6)Nihongaki, Y. et al. Photoactivatable CRISPR-Cas9 for optogenetic genome editing. Nat. Biotechnol., 33, 755–760 (2015).
7)Nihongaki, Y. et al. CRISPR–Cas9-based photoactivatable transcription systems to induce neuronal differentiation. Nat. Methods, 14, 963–966 (2017).
8)Konermann, S. et al. Genome-scale transcriptional activation by an engineered CRISPR-Cas9 complex. Nature, 517, 583–588 (2015).
9)Tak, Y. E. et al. Inducible and multiplex gene regulation using CRISPR-Cpf1-based transcription factors. Nat. Methods, 14, 1163–1166 (2017).
<お問い合わせ先>
<研究に関すること>
佐藤 守俊(サトウ モリトシ)
東京大学 大学院総合文化研究科 教授
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構 戦略研究推進部 ライフイノベーショングループ
<報道担当>
科学技術振興機構 広報課



