2022-10-25 アメリカ・ローレンスリバモア国立研究所(LLNL)
この研究は、宇宙ステーションの環境プロファイル(マイクロバイオーム)の初めての包括的な特性評価であり、メタゲノムDNA配列決定技術を使用してISSのマイクロバイオームと宇宙飛行士のマイクロバイオームを比較した最初のものである。
2015年から2020年までの5年間の研究期間中、研究チームは2つの主要な研究を行った。Venkateswaren氏が率いる微生物追跡(MT)-1プロジェクトでは、宇宙ステーションの表面と空気サンプルから得られた微生物の多様性が、従来の微生物学と分子技術の両方を用いて特性評価された。aing氏が率いるMT-2プロジェクトでは、MT-1プロジェクトで調査したものと全く同じ表面から採取した宇宙飛行士のサンプルを比較し、最初の調査をさらに発展させた。
ISSの抗菌性遺伝子プロファイルも、MT-1とMT-2の研究期間中に差はなく、経時的に安定していました。これは、ISSのマイクロバイオームには新しい抗生物質耐性遺伝子がないことを意味し、宇宙飛行士にとってより安全であることを意味する。
宇宙飛行士と宇宙ステーションの環境の両方から採取したサンプルの微生物組成は、メタゲノム配列を用いて測定され、膨大な量のDNA配列データから細菌、ウイルス、真菌などの微生物を迅速に特定するバイオインフォマティクスソフトウェア、リバモアメタゲノミクス解析ツールキットを用いて処理された。
<関連情報>
- https://www.llnl.gov/news/lab-microbial-array-used-space-station-study
- https://microbiomejournal.biomedcentral.com/articles/10.1186/s40168-022-01293-0
国際宇宙ステーション搭載の細菌・真菌のメタゲノム解析「Microbial Tracking-2(マイクロビアントラッキング-2)」。 Microbial Tracking-2, a metagenomics analysis of bacteria and fungi onboard the International Space Station
Camilla Urbaniak,Michael D. Morrison,James B. Thissen,Fathi Karouia,David J. Smith,Satish Mehta,Crystal Jaing & Kasthuri Venkateswaran
Microbiome Published:29 June 2022
DOI:https://doi.org/10.1186/s40168-022-01293-0
Abstract
Background
The International Space Station (ISS) is a unique and complex built environment with the ISS surface microbiome originating from crew and cargo or from life support recirculation in an almost entirely closed system. The Microbial Tracking 1 (MT-1) project was the first ISS environmental surface study to report on the metagenome profiles without using whole-genome amplification. The study surveyed the microbial communities from eight surfaces over a 14-month period. The Microbial Tracking 2 (MT-2) project aimed to continue the work of MT-1, sampling an additional four flights from the same locations, over another 14 months.
Methods
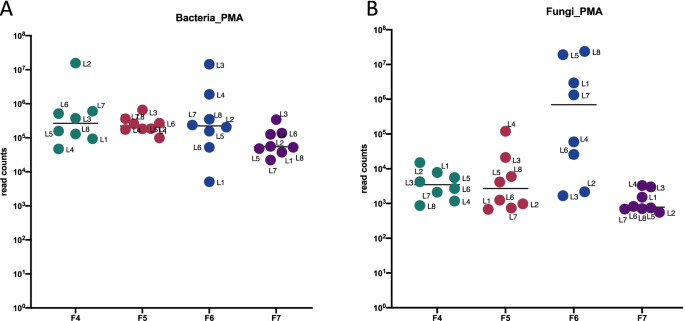
Eight surfaces across the ISS were sampled with sterile wipes and processed upon return to Earth. DNA extracted from the processed samples (and controls) were treated with propidium monoazide (PMA) to detect intact/viable cells or left untreated and to detect the total DNA population (free DNA/compromised cells/intact cells/viable cells). DNA extracted from PMA-treated and untreated samples were analyzed using shotgun metagenomics. Samples were cultured for bacteria and fungi to supplement the above results.
Results
Staphylococcus sp. and Malassezia sp. were the most represented bacterial and fungal species, respectively, on the ISS. Overall, the ISS surface microbiome was dominated by organisms associated with the human skin. Multi-dimensional scaling and differential abundance analysis showed significant temporal changes in the microbial population but no spatial differences. The ISS antimicrobial resistance gene profiles were however more stable over time, with no differences over the 5-year span of the MT-1 and MT-2 studies. Twenty-nine antimicrobial resistance genes were detected across all samples, with macrolide/lincosamide/streptogramin resistance being the most widespread. Metagenomic assembled genomes were reconstructed from the dataset, resulting in 82 MAGs. Functional assessment of the collective MAGs showed a propensity for amino acid utilization over carbohydrate metabolism. Co-occurrence analyses showed strong associations between bacterial and fungal genera. Culture analysis showed the microbial load to be on average 3.0 × 105 cfu/m2
Conclusions
Utilizing various metagenomics analyses and culture methods, we provided a comprehensive analysis of the ISS surface microbiome, showing microbial burden, bacterial and fungal species prevalence, changes in the microbiome, and resistome over time and space, as well as the functional capabilities and microbial interactions of this unique built microbiome. Data from this study may help to inform policies for future space missions to ensure an ISS surface microbiome that promotes astronaut health and spacecraft integrity.