2019-11-18 理化学研究所
理化学研究所(理研)生命機能科学研究センター計算分子設計研究チームの泰地真弘人チームリーダーらの研究チーム※は、分子動力学(MD)[1]シミュレーション専用計算機「MDGRAPE-4A[2]」の開発に成功しました。
本研究成果は、インシリコ創薬[3]の可能性を大きく拡げるものと期待できます。
MDシミュレーションは、水溶液中で変化し続けるタンパク質構造を解析するために、タンパク質を構成する原子や周囲の水分子に働く力を計算し、コンピュータ内でタンパク質を「動かす」手法です。大きなタンパク質の解析には、汎用スーパーコンピュータ[4](スパコン)でも膨大な時間がかかるため、分子シミュレーションを高速で行う専用スパコンの開発が待たれていました。
今回、研究チームは、自ら設計・開発した専用の大規模集積回路(LSI)[5]を512個搭載し、システム全体として約1.3ペタフロップス(1秒間に1,300兆回)の計算能力を持つMDGRAPE-4Aを開発しました。MDGRAPE-4Aは、タンパク質と水分子からなる10万原子系[6]のシミュレーションを、1日の計算で最高1.1マイクロ秒(1マイクロ秒は100万分の1秒)進める性能を持ちます。これにより、サブミリ秒(~100マイクロ秒)のタイムスケールで起きる水溶液中でのタンパク質と薬剤の分子間相互作用の解析が、現実的な時間で可能となります。
本開発は、東京都で開催された「情報計算化学生物学会2019年大会」(10月24日)で報告されました。また、完成品の一部を米国デンバーで開催される国際会議「Supercomputing SC19」(現地時刻11月18日~21日)で展示し、システム全体は理研大阪地区一般公開2019(11月23日)で公開します。
背景
細胞のさまざまな機能は、タンパク質などの分子が相互作用することにより制御されています。この仕組みを利用した分子標的薬[7]の開発では、がん細胞や病原体が持つ標的タンパク質に結合し、その機能を阻害する化合物を探索することが基本になります(図1)。しかし、生体内(溶液中)のタンパク質の構造は柔らかく、ゆらゆらと常に変化しています。このような構造変化は、X線結晶解析[8]など通常の構造解析の手法で解析することは困難です。そこで近年、スーパーコンピュータ(スパコン)を用いた分子シミュレーションにより標的タンパク質の構造変化を再現し、それに結合する化合物の候補を膨大な仮想化合物ライブラリーの中からスクリーニングする「インシリコ創薬」が注目されています。
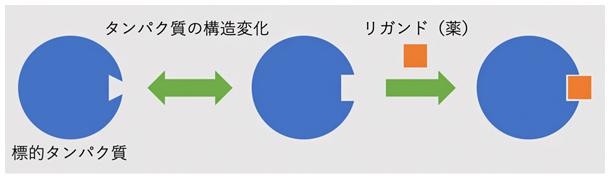
図1 分子標的薬がタンパク質に作用する仕組み
薬となる化合物(リガンド)は、体内で特定のタンパク質に強く結合することで、その機能を阻害するなどの効果を発揮する。
タンパク質の構造変化のシミュレーションでは、タンパク質を構成する数千個以上の原子間に働く力や、これらの原子とタンパク質を取り囲む数万個の水分子との間に働く力を、時間刻みで計算します。この計算結果から全ての原子の動きを求めることを繰り返すことで、タンパク質全体の動きをあたかも映画のコマを1コマ1コマ進めるようにシミュレーションします。これは分子動力学(MD)計算という手法で(図2)、膨大な時間がかかるのが特徴です。
生体分子の計算では、2フェムト秒(2×10-15秒)程度の動きを1コマとして計算します。生体内でのタンパク質の大規模な構造変化は、マイクロ秒(100万分の1秒)からミリ秒(1000分の1秒)、あるいはそれ以上のタイムスケールで起きると考えられており、例えば1コマ2.5フェムト秒(2.5×10-15秒)で10万原子系の100マイクロ秒間(1×10-4秒)の動きを再現するには、400億(4×1010)コマの計算が必要となります。現行の汎用スパコンでは1コマあたりの計算に最短でも約1ミリ秒(1×10-3秒)秒かかるため、400億コマの計算に必要な時間は最短で4000万秒(4×107秒、約1年3カ月)となります。
スパコンの性能が上がっても、1コマあたりの時間をミリ秒以下にすることは汎用計算を行う設計上困難であるため、分子シミュレーションを高速で行う専用スパコンの開発が待たれていました。
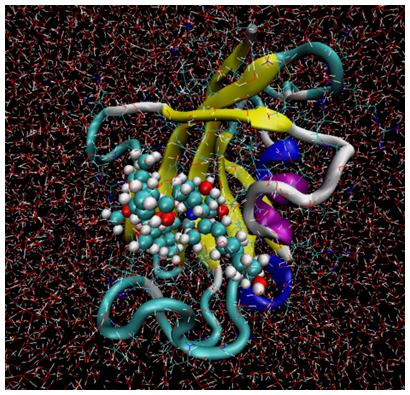
図2 分子動力学(MD)計算を用いた高精度シミュレーションの例
図はMD計算で得られたタンパク質の高精度シミュレーションの例。リボンモデルで表した分子はタンパク質を、ボール&スティックモデルで表した分子はリガンドを示す。計算によって結合しやすさを求める際、実際にはタンパク質も薬の分子も形を変えるため、その変化をとらえることが重要である。MD計算では、タンパク質や薬となる分子、周囲の水分子を原子レベルでモデル化し、その時間発展を追う。
研究手法と成果
研究チームは、MD計算で必要な粒子間の力の計算に特化した加速装置を大型集積回路(LSI)に組み込み、このLSIを512個実装したMDシミュレーション専用スパコン「MDGRAPE-4A」を開発しました(図3)。MDGRAPE-4Aでは、タンパク質と水分子からなる10万原子系のシミュレーションを、1日の計算で最高1.1マイクロ秒間進める性能を持ちます。これにより、100マイクロ秒間の動きに必要な計算時間は91日間となり、汎用スパコンで最短でも1年3カ月かかっていたシミュレーションを約3カ月で完了することができます。
これまで理研が開発してきた専用計算機では、計算の一部のみを専用化し、残りは通常のコンピュータで計算していました。しかし、専用計算機が高速になるにつれ、この方式では通常のコンピュータの部分で性能が頭打ちになるようになってきました。そこで、MDGRAPE-4Aではこれまでの専用計算回路に加え、汎用計算部分やネットワークなど計算の全てを一つのLSIに統合した大規模な「システムオンチップ(SoC)[9]」とすることで、ボトルネックの解消を図っています。この実現には、多くの新しい技術開発が必要となりました。主要なものとしては、以下が挙げられます。
(1)遠くの原子間に働く力の計算を加速するための、専用計算機に適した計算アルゴリズムの開発とハードウェア実装。
(2)近くの原子間で働く力を計算する高速の専用回路。
(3)512個のLSIを光ファイバーでつなぐ高速・低遅延のネットワーク。
(4)RISC-V[10]をMD計算向けに変更したプロセッサ。
(5)演算器やデータ管理回路を埋め込んだメモリ。
(6)FPGA(プログラム可能な集積回路)[11]による超高速な3次元FFT[12]の実装。
こうした個別の工夫に加え、LSIに実装された多くの要素全てを連携させて高速に動作できるよう、ハードウェアとソフトウェアの共設計を進めました。さらに512個のLSIをシステムとして組み上げることにより、システム全体として約1.3ペタフロップス(1秒間に1,300兆回)の計算能力を持ち、高速に計算を行うことができるシステムの動作を達成しました。本システムは、RISC-Vをベースとした実用大規模システムとしては世界初です。

図3 今回開発したMDGRAPE-4Aシステム
MD専用計算機の開発では、チップ上にメモリ(GM)、汎用プロセッサ(GP)、粒子間相互作用パイプライン(PP)、ネットワークインターフェース(NIF)を統合したLSIを独自に設計した。左側4列のように、LSIを8個搭載した基板を光接続とFPGAシステムによって64枚連結し、全体で一つのMD計算を高速に行う。
今後の期待
インシリコ創薬の技術は、候補分子の構造式を用いて実施できるという大きな利点を持ち、ほとんど無制限ともいえる数の化学構造式をスクリーニングの対象にできます。MDGRAPE-4Aによる長時間シミュレーションを実行すれば、候補分子とタンパク質とが実際に結合するときの構造変化を探索し、より高精度な予測が実現できます。また本計算手法は、タンパク質の「形」だけではなく、「動き」を制御する分子を開発する上でも有望であり、創薬の可能性を大きく広げるものと期待できます。さらに将来的には、創薬以外の広い分野での産業・アカデミアへの共用に供していきたいと考えています。
計算機開発の面では、半導体の性能向上を支えてきた「ムーアの法則[13]」が終焉を迎える中、専用計算システムの役割がますます重要になると考えられます。MD計算において、ハードウェアとソフトウェアの両面で「深いレベルの統合」を行った開発を現在進めているのは、米国のD. E. Shaw研究所[14]と理研の本研究チームのみであり、今後もMD計算のさらなる加速に加え、人工知能への応用等、専用回路と汎用回路の結合による大規模システムの開発を推進していきます。
補足説明
1.分子動力学(MD)
原子間に働く力を計算し、運動方程式を繰り返し解くことで、分子の動きを追跡する方法。分子動力学法の基礎の開発について、2013年のノーベル化学賞が授与されている。MDはMolecular Dynamicsの略。
2.MDGRAPE-4A
1990年より開発が進められている天文学分野での重力(GRAvity)多体問題の計算に特化した専用計算機GRAPE(GRAvity PipE、重力パイプライン)の、分子動力学(Molecular Dynamics: MD)バージョン。MDGRAPE-4Aはその5作目に当たる。なお、MDGRAPE-3は高性能計算科学の賞であるゴードンベル賞を受賞している(2006年)。
3.インシリコ創薬
細胞生物学的、生化学的な手法を主とする創薬候補物質の探索に対して、コンピュータ(シリコンチップ)の中で行う創薬をインシリコ(in silico)創薬と呼ぶ。
4.汎用スーパーコンピュータ
さまざまな用途で高速計算を行えるコンピュータ。スーパーコンピュータ「京」やその後継機「富岳」は、汎用スーパーコンピュータの例。
5.大規模集積回路(LSI)
特定の演算機能を持たせるために、多数の素子を一つにまとめた電子部品を集積回路(Integragted Circuit, IC)といい、素子の集積度が1,000個~10万個程度のものを大規模集積回路(LSI)と呼ぶ。LSIはLarge Scale Integrationの略。
6.原子系
物質を、力学法則に従う粒子(原子)の集合として捉えた系。水溶液中の一般的なタンパク質は、タンパク質を構成する数千個の原子と、周囲の数万個の水分子からなる10万原子系と見なすことができる。
7.分子標的薬
特定の疾患に関与する分子だけに作用する薬剤。例えばがん治療においては、増殖中の細胞が一般的に持つ性質を利用する従来の抗がん剤に対して、がん細胞の増殖に特に関与する分子を標的とする薬剤を開発することにより、副作用の軽減が期待される。
8.X線結晶解析
構造生物学の手法の一つ。タンパク質の結晶を作製し、その結晶にX線を照射して得られる回折データを解析することにより、タンパク質の内部の原子の立体的な配置を調べる方法。この方法によって、タンパク質の形(立体構造)や内部構造を知ることができる。
9.システムオンチップ(SoC)
演算処理を担うCPUのみを集積した回路に対し、メモリや信号処理を含めたシステム全体を一つのチップに載せたもの。
10.RISC-V
カリフォルニア大学バークレイ校を中心に開発されているコンピュータの命令セットアーキテクチャ(Instruction Set Architecture: ISA)の一つ。RISC-Vは完全にオープンであり、RISC-VのISA使うためのライセンス料は不要。
11.FPGA(プログラム可能な集積回路)
製造後に購入者や設計者が構成を設定できる集積回路。FPGAはField-Programmable Gate Arrayの略。
12.3次元FFT
計算機上で離散フーリエ変換を高速に計算するアルゴリズム。信号処理などで頻繁に用いられている。例えば2次元FFTは、よく使われている「JPEG」フォーマットでの画像データ圧縮の基礎である。FFTは、Fast Fourier Transform(高速フーリエ変換)の略。
13.ムーアの法則
世界最大の半導体メーカーIntel社の創設者の一人であるGordon Moore博士が1965年に経験則として提唱した、「半導体の集積密度は18~24カ月で倍増する」という法則。
14.D. E. Shaw研究所
計算機科学研究者でありヘッジファンド創設者のD. E. Shawが創設した米国の私設研究所。分子シミュレーション専用計算機の開発に際して、集積化したチップの搭載を世界に先駆けて実現した。
参考 MDGRAPE-4Aシステム諸元
専用LSI:MDGRAPE-4A SoC
プロセスノード:40nm
チップ面積:16.28mmx16.28mm
動作クロック:600/800MHz
メモリ:2メガバイト オンチップメモリ
専用パイプライン:64本
長距離演算ユニット:2個
汎用演算コア(拡張RISC-V):16+1個
32ビット固定小数点ベクトル命令等の拡張演算:64キロバイト局所メモリ
3次元トーラスネットワーク:双方向7.2ギガバイト/秒 (計6Gbps 72レーン)
MDGRAPE-4Aシステム
8個x8個x8個(計512個)のSoCを3次元トーラスネットワークで接続
1ボードに8個のSoCを実装
ボード間接続には光接続を利用
長距離力専用FPGAシステム(計9個のFPGAにより構成)
消費電力:65kW(推定値)
冷却システム:ヒートシンク・ファンによる空冷
開発費:約7億円(研究者の人件費を除く)
開発期間:約2年間
設置場所:理研大阪地区(大阪府吹田市)
研究チーム
理化学研究所
生命機能科学研究センター
計算分子設計研究チーム
チームリーダー 泰地 真弘人(たいじ まこと)
上級技師 大野 洋介(おおの ようすけ)
技師 森本 元太郎(もりもと げんたろう)
研究員 小松 輝久(こまつ てるひさ)
研究員 小山 洋平(こやま ようへい)
研究員 大村 一太(おおむら いった)
研究員 チョウ コウ(Zhang Hao)
大学院生リサーチ・アソシエイト 西田 圭吾(にしだ けいご)
制御分子設計研究チーム
チームリーダー 本間 光貴(ほんま てるき)
創薬・医療技術基盤プログラム
プログラムディレクター 後藤 俊男(ごとう としお)
連携促進コーディネーター 小山 洋(こやま ひろし)
研究支援
本開発は日本医療研究開発機構(AMED)創薬支援推進事業-創薬支援インフォマティクスシステム構築、日本学術振興会(JSPS)最先端研究基盤事業「生命動態システム科学研究の推進(実施機関:理化学研究所)、科学研究費補助金基盤研究(A)「次世代分子動力学シミュレーション専用計算機の基盤開発(研究代表者:泰地真弘人)」から一部支援を受け、大正製薬株式会社との共同研究により実施したものです。
LSI開発の一部は、アルチップ・テクノロジーズ株式会社、ディー・クルー・テクノロジーズ株式会社、東京エレクトロン デバイス株式会社、株式会社クロスアビリティへの委託のもと行われました。システムの設計・製造にあたっては、東京エレクトロン デバイス株式会社への委託のもと、株式会社ホックス、インテグラン株式会社、株式会社フジクラの協力を得ました。
原論文情報
タイトル(発表者名)
MDGRAPE-4Aシステムの概要とMDGRAPE-5への展望(泰地 真弘人)
MDGRAPE-4Aシステムの運用と性能評価(森本 元太郎、小松 輝久)
インシリコスクリーニング活性予測の現状とMDGRAPE-4Aへの期待(本間 光貴)
学会
情報計算化学生物学会2019年大会
特許
特願2018-197597 発明の名称:処理装置、処理システム、処理方法、プログラム、及び、記録媒体
発表者
理化学研究所
生命機能科学研究センター 計算分子設計研究チーム
チームリーダー 泰地 真弘人(たいじ まこと)
お問い合わせ先
理化学研究所 生命機能科学研究センター センター長室 報道担当
山岸 敦(やまぎし あつし)
報道担当
理化学研究所 広報室 報道担当