ニホンナシにおけるゲノムワイド関連解析とゲノミックセレクション
- 2018/08/15 東京大学,農研機構
- 発表者
- 南川 舞(東京大学大学院 農学生命科学研究科 生産・環境生物学専攻 特任研究員)
髙田 教臣(農研機構果樹茶業研究部門 品種育成研究領域 主任研究員:当時)
寺上 伸吾(農研機構果樹茶業研究部門 品種育成研究領域 主任研究員)
齋藤 寿広(農研機構果樹茶業研究部門 品種育成研究領域 ユニット長)
小野木 章雄(東京大学大学院 農学生命科学研究科 JSTさきがけ専任研究員:当時)
鐘ケ江 弘美(東京大学大学院 農学生命科学研究科 生産・環境生物学専攻 特任助教)
林 武司(農研機構次世代作物開発研究センター 基盤研究領域 ユニット長)
山本 俊哉(農研機構果樹茶業研究部門 品種育成研究領域 ユニット長)
岩田 洋佳(東京大学大学院 農学生命科学研究科 生産・環境生物学専攻 准教授)
発表のポイント
◆実際の育種集団を活用し、果樹育種の効率化に貢献するゲノムワイド関連解析(GWAS)とゲノミックセレクション(GS)の枠組みを提唱しました。
◆収穫期、果実重、果汁酸度などに関連する遺伝子候補領域を検出し、それらの特性を高い精度で予測できることを示しました。また、複数の果実特性を同時に予測する手法の有効性を確認しました。
◆今回提唱したGWAS、GSの枠組みは、ニホンナシにおける育種効率の向上に寄与するだけでなく、他の果樹育種への応用も可能です。
発表概要
近年、ゲノムワイドマーカー(注1)を利用した、GWAS(注2)やGS(注3)などのゲノム育種法が注目されています。GWASやGSを成功させるためには、非常に多くのデータセット(マーカー遺伝子型と表現型情報)を用意する必要がありますが、果樹は、世代の期間が長く、果実特性の評価に多大なコストを要するため、データを収集するのが容易ではありません。
東京大学と農研機構の研究グループは、育種の現場で実際に育成されているニホンナシの品種・系統に加えて複数の交配育種集団の情報を活用することで、高精度なGWASやGSが可能であることを証明しました。収穫期、果実重、果汁酸度などで、GWASで遺伝子候補領域の検出に成功し、高精度でのGS予測が可能であることを示しました。また、複数の果実特性を同時に予測する手法の有効性も確認しました。
今後、多数の交配育種集団の情報を蓄積していくことで、GWASとGSの精度をさらに向上できる可能性があります。今回提唱したGWAS、GSの枠組みは、ニホンナシにおける育種効率の向上に寄与するだけでなく、他の果樹育種への応用も期待されます。
発表内容
【研究の背景】
果樹は一般的に種子が発芽してから開花・結実するまでの期間が長く、交配育種に長い年月を要します。また、個体サイズが大きいために多数の個体を選抜対象にできず、新品種の育成が容易ではありません。ニホンナシの場合、交配で得られる個体から新しい品種が生まれる確率は、1000個体に1個体程度です。近年注目されているGWASやGSなどのゲノム情報を活用した解析や育種法は、果樹育種のこうした障壁を取り除く可能性があります。しかし、GWASやGSを成功させるためには、非常に多くのデータセット(マーカー遺伝子型と表現型情報)を用意する必要があり、上記のような障壁を持つ果樹は、どのようにデータを収集するべきかが課題でした。そこで本研究では、農研機構の育種の現場で実際に育成されているニホンナシ品種・系統と交配育種集団を用いて、GWASとGS予測精度評価を行い、ゲノム育種の可能性評価を行いました。
【研究内容】
ニホンナシ86品種・系統と、16きょうだい集団(注4)から構成される交配育種集団から得られた1,506個のゲノムワイドマーカーおよび、18の特性(14果実特性、2病害抵抗性、2成長特性)の表現型情報を解析に用いました。品種・系統のみの場合と品種・系統と交配育種集団を統合した場合とでGWASを行い、結果を比較しました。また、3つの異なるパターンで構成されたトレーニング集団を用いてGS予測モデルを構築し、交配育種集団の表現型予測精度を評価しました。また、果実特性間の遺伝相関の情報を考慮して、複数の果実特性を同時に予測することができる多形質モデルの検討も行いました。予測値と実際の観察値の相関(r)の大きさを予測精度の指標としました。
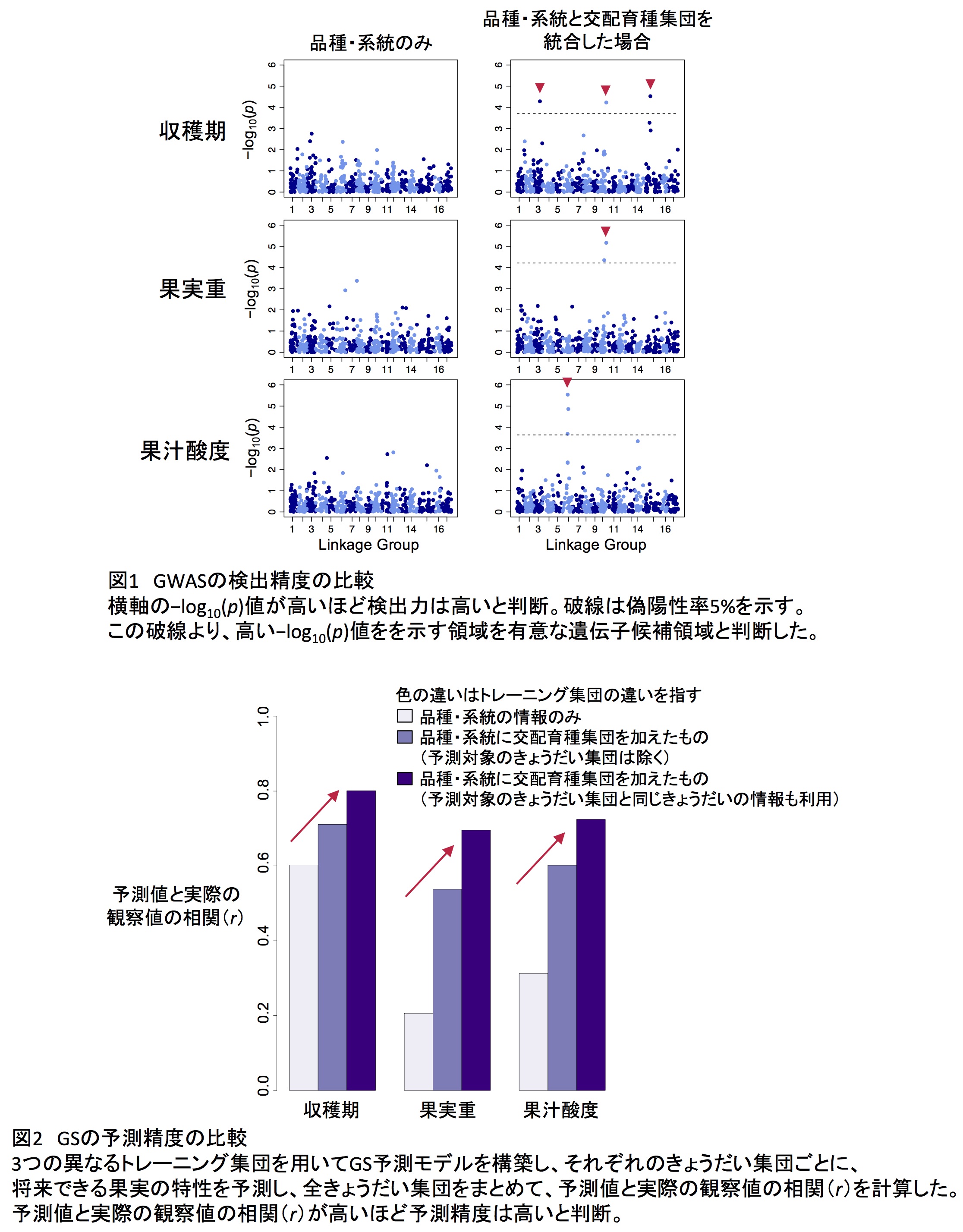
品種・系統と交配育種集団を統合した場合のGWASでは、品種・系統のみを用いた場合のGWASに比べて多数の有意な遺伝子候補領域が検出され、収穫期、果実重、果汁酸度などでは新規の遺伝子候補領域が存在する可能性を示しました(図1)。また、品種・系統のみよりも、品種・系統と交配育種集団を統合した集団(予測されるきょうだい集団は除く)をトレーニングデータとしてモデルを構築した場合に、交配育種集団の表現型予測精度が高く、トレーニングデータに予測対象のきょうだい集団と同じきょうだいの情報も含めることで予測精度はさらに向上しました(図2)。通常、初めての交配組み合わせで生まれた予測対象のきょうだい集団の情報を利用することはできません。GSは芽生えの段階で行うため、予測対象のきょうだい集団の果実特性の情報を集めることができないからです。しかし、有望な個体が得られた場合には、より優秀な個体の獲得を目指して、同じきょうだいの個体を追加で調査することがあります。この場合には、予測対象のきょうだい集団と同じきょうだいの情報をトレーニングデータとして利用可能となります。多形質モデルでは、それぞれの果実特性ごとのモデルで予測精度の低かった、収穫前落果率やみつ症率について予測精度が向上しました。
以上の結果より、データ収集が難しい果樹において、GWASやGSに実際の育種集団の情報を活用することが有効であることがわかりました。また、多形質モデルを使用することで、それぞれの果実特性ごとに予測モデルを構築する必要がなく、GSの効率性を向上できる可能性を示しました。今後、交配育種を行いながら多数の交配育種集団のマーカー遺伝子型と表現型情報を蓄積していくことで、GWASとGSの精度をさらに向上できる可能性があります。
【社会的意義・今後の予定】
今回提唱した、実際の育種集団の情報を活用するGWAS、GSの枠組みは、ニホンナシにおける育種効率の向上に寄与するだけでなく、他の果樹育種への応用も期待されます。
今後は、より高精度なGWASとGSを目指して、マーカー遺伝子型と表現型情報を蓄積するとともに、より簡便にデータを蓄積できるようなデータベースシステムの構築も必須であると考えられます。また、果実特性については、客観的でより高精度な評価方法を検討する必要があります。
なお、本研究は農林水産省委託プロジェクト「ゲノム情報を活用した農産物の次世代生産基盤技術の開発」の支援を受けて行われました。
発表雑誌
- 雑誌名:Scientific Reports
- 論文タイトル:Genome-wide association study and genomic prediction using parental and breeding populations of Japanese pear (Pyrus pyrifolia Nakai)
- 著者:Mai F. Minamikawa, Norio Takada, Shingo Terakami, Toshihiro Saito, Akio Onogi, Hiromi Kajiya-Kanegae, Takeshi Hayashi, Toshiya Yamamoto, Hiroyoshi Iwata*
- DOI番号:10.1038/s41598-018-30154-w
- 論文URL
- :http://www.nature.com/articles/s41598-018-30154-w
問い合わせ先
東京大学大学院 農学生命科学研究科 生産・環境生物学専攻 生物測定学研究室
准教授 岩田 洋佳 (いわた ひろよし)
用語解説
- 注1 ゲノムワイドマーカー
- ゲノム全体に配置した高密度なDNAマーカーです。DNAマーカーとは生物個体の遺伝的性質、もしくは系統(個人、親子・親族関係、血統あるいは品種など)を特定するための目印となる個体特有のDNAのことです。
- 注2 ゲノムワイド関連解析(GWAS)
- 多数の品種・系統におけるDNAの違い(マーカー遺伝子型)と果実特性(表現型)の関係を数式によってモデル化し、果実特性と関連するDNAの違いを統計的に検出する手法です。果実特性と関連するDNAの違いが明らかになれば、そのDNAの近傍を探索することで、果実特性を支配する候補遺伝子を同定することができます。
- 注3 ゲノミックセレクション(GS)
- DNAの違いの情報をもとに、個体の遺伝的能力を予測して選抜する方法です。DNAの違いと果実特性のデータについて、多数の品種・系統をトレーニングデータとして両者間に見られる関係を数式によってモデル化し、その「予測モデル」に基づいて、個体の遺伝的能力を予測します。実生苗の段階で将来できる果実の性質を予測することが可能です。
- 注4 きょうだい集団
- 一つの交配組み合わせから生まれた、同じ両親を持つきょうだいの集まりを、きょうだい集団と呼んでいます。