2022-11-17 広島大学
研究成果のポイント
- 本研究グループでは、公共データベースを利活用してミツバチのゲノム配列解析を行ってきましたが、再現性のある遺伝子配列データ解析をはじめとしたデジタルトランスフォーメーション(バイオDX)のためにはきちんとした遺伝子配列セットが必要とされてきました。
- そこで今回、公共データベース中から利用可能なセイヨウミツバチの遺伝子発現データを専門家の目によって選び抜き、該当するデータをすべて再解析することによって網羅的な参照遺伝子セットとして構築しました。
- 今回構築した参照遺伝子セットを利用して、感染症に関連したセイヨウミツバチの遺伝子発現データの再解析を行い、新規の免疫関連候補遺伝子を複数同定しました。この解析手法は、今回の解析事例のみならずさまざまな生物学的現象に関係する遺伝子群のデータ駆動的な探索手法として今後活用できると期待されます。
概要
広島大学大学院統合生命科学研究科の若宮健研究員(当時)と坊農秀雅特任教授、農研機構の横井翔主任研究員の研究グループは、公共遺伝子発現データからミツバチの参照遺伝子セットを構築し、それを用いたトランスクリプトームデータ解析手法を構築しました。
ミツバチは蜂蜜の生産や施設栽培の野菜や果物における花粉媒介に不可欠な経済的に重要な昆虫種です。そのため、ミツバチの代表的な種であるセイヨウミツバチ(Apis mellifera)を中心に昆虫学的な研究において幅広く研究されてきました。バイオデジタルトランスフォーメーション(バイオDX)は昆虫を含む様々な生物種における遺伝子情報などの生物に関するデジタルデータを駆使した解析を意味します。この解析の中には遺伝子配列データ解析などが含まれます。これらの解析によって、これまでにない様々な有用な生物機能をゲノム編集で付与することが可能になると期待されています(例えば病気に強いミツバチなど)。再現性のある遺伝子配列データ解析をはじめとしたバイオDXを推進するためには、基準となる参照遺伝子セットが必要不可欠です。
本研究では、公共データベース中から利用可能なセイヨウミツバチの遺伝子発現データをすべて再解析することによって、149,685種類の転写産物(16,735遺伝子由来)からなる参照転写産物データセット(参照トランスクリプトームデータセット)を構築しました。複数のモデル生物や昆虫種のタンパク質配列データセットを用いて、構築したタンパク質配列の約半分に機能情報を付与(アノテーション)することができました。
構築した参照転写産物データセットの有用性を確認するため、公共データベースにあるトランスクリプトームデータセットを利用した新規の免疫反応関連遺伝子の探索に用いました。その結果、ウイルスとバクテリアの感染に反応して発現が変動する遺伝子を複数同定しました。このメタ解析の結果、参照遺伝子セットは、セイヨウミツバチのトランスクリプトーム解析に使用できることが示されました。この研究で用いられた解析手法は、ミツバチの感染症の研究のみならず他の研究分野にも展開可能な手法と考えています。
本研究成果はスイスの出版社 Multidisciplinary Digital Publishing Institute (MDPI)の Insects 誌に10月14日に掲載されました。
論文情報
著者
Kakeru Yokoi, Takeshi Wakamiya, Hidemasa Bono*
* Corresponding author(責任著者)
論文題目
Meta-analysis of the public RNA-Seq data of the Western honeybee Apis mellifera to construct reference transcriptome data
掲載雑誌
Insects
DOI: 10.3390/insects13100931
発表内容
【研究の背景】
ミツバチは経済的にも生態的にも重要な昆虫種です。蜂蜜の生産に不可欠なだけでなく、農作物と野生植物の両方の受粉を媒介する昆虫です。また、社会性昆虫のモデル種として、昆虫学の研究分野においても長年研究されています。とくにセイヨウミツバチ(Apis mellifera)は、生物学的研究、受粉媒介、蜂蜜生産等に広く応用されている代表的なミツバチ種です。その有用性から、ゲノム配列解読が古くからなされ、2006年にその参照ゲノム配列データが公開されました。参照ゲノム配列データは継続的に更新され、DNA配列決定技術の進歩に伴い、2019年に染色体レベルのゲノム配列データが公開されました。セイヨウミツバチのゲノムおよびトランスクリプトームデータを用いた研究によって、分子および遺伝学的証拠に基づく新しい洞察が発表されたことなどもあり、RNA-Seqによるトランスクリプトーム解析が進められてきています。
【研究成果の内容】
本研究では、公共データベース中から利用可能なセイヨウミツバチの遺伝子発現データをすべて再解析することによって、149,685の遺伝子配列と194,174のタンパク質配列からなる参照遺伝子セットを構築しました(図1)。複数のモデル生物や昆虫種のタンパク質配列データセットを用いて、構築したタンパク質配列の約半分に機能情報を付与(アノテーション)することができました。
さらに、構築した参照遺伝子セットの有用性を確認するため、公共データベースにあるミツバチの感染症に関連するトランスクリプトームデータセットを利用した新規の免疫反応関連遺伝子の探索に用いました(図1)。その結果、ウイルスとバクテリアの感染に反応して発現が増加または減少する遺伝子を3-20個同定しましたが、その中にはこれまで昆虫において免疫に関連すると知られている遺伝子は含まれていませんでした。また、オートファジー関連タンパク質3は、ウイルス感染によって発現が減少いることが確認されましたが、このことはオートファジーがウイルス性免疫反応に関与していることを示唆しています。
以上の解析の結果から、参照遺伝子セットはセイヨウミツバチのトランスクリプトーム解析に使用できることが示されました。この研究で用いられた解析手法は、ミツバチの感染症の研究のみならず他の様々な研究分野にも展開可能な手法と考えています。
【今後の展開】
開発した解析手法は、ミツバチの研究における有用なデジタルリソースとなり、ゲノム編集を利用した有用形質を付与したミツバチの作出につながると期待されます。また、ミツバチばかりでなく、農業や畜産に重要な生物のゲノム編集に向けてその遺伝子発現データを解析する際の強力なデータ解析手段となることが期待されます。
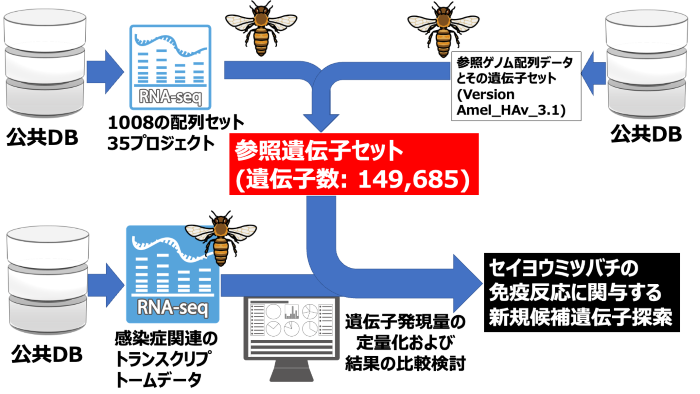
図1 本研究の全体図
用語説明
※参照配列:ここでは転写産物の参照配列のことを指す。各転写産物の発現量を定量するために参照する配列のこと。
※RNA-seq:サンプル中の検出可能なすべての遺伝子について、その配列の解析や発現量を網羅的に測定できる技術。
対象とするSDGs:
SDGs 3 すべての人に健康と福祉を
SDGs 9 産業と技術革新の基盤をつくろう
【お問い合わせ先】
<研究に関すること>
大学院統合生命科学研究科 特任教授 坊農秀雅
<報道に関すること>
広島大学広報室