NREL主導の研究チームは、機械学習を用いて、あらゆる種類のPET(耐久性のある結晶性のもの)を分解するのに適した「プラスチック分解酵素」の変種を発見しました。 Using Machine Learning, NREL-Led Team Discovered Variants of ‘Plastic-Eating’ Enzymes Adapted To Deconstructing All Varieties of PET—Even Durable Crystalline Varieties
2022-12-21 米国国立再生可能エネルギー研究所(NREL)
Nature Communications』誌に掲載された論文によると、ポーツマス大学とモンタナ州立大学のベッカム教授らは、こうした方法を用いて新しい酵素の変異体を発見し、余分な前処理をせずに最も頑丈なPETを分解できる可能性があることを明らかにしました。
PETの酵素リサイクルという概念は2005年から知られていましたが、2016年に日本の科学者が驚くべき発見をしたことから、世界の舞台で急成長しました。日本のリサイクル施設の外の土に埋もれて、彼らがIdeonella sakaiensisと名付けた細菌が静かに酵素を分泌し、散乱した古い飲料用ペットボトルを分解していたのです。
その後、さまざまな研究が行われた。毎年何百万トンも生産されるPETボトルを処理するための工業技術として、この酵素を改良しようとした。
しかし、急速な進歩にもかかわらず、工業規模の酵素リサイクルには重要な障壁が立ちはだかった。
酵素が効果を発揮するのは、ごく一部のPET製品、つまり「非結晶性」PETから作られたものだけだったのだ。一般的で耐久性の高い「結晶性」PETを分解するには、まず高熱と余分なエネルギーで軟化させる必要があり、苦労していた。結晶性PETは、衣類に使われるポリエステル繊維や使い捨ての飲料用ボトルの部品など、生産されるPETの約90%を占めている。
そこで研究者たちが必要としたのは、結晶性PETの分解をより得意とする酵素だった。
研究チームは新しい酵素の種類を探すのに、バイオインフォマティクスと機械学習の進歩により、既存の酵素配列の膨大なデータベースから、結晶性PETに作用する品種を探し出すことがすでに可能になっていた。
この課題を解決するため、我戸は既知のプラスチック分解酵素の生物学的法則を学習する統計モデルを構築した。このモデルは、これまでに研究された酵素のユニークな構成に確率を割り当てた。さらに、産業用途で重要な酵素の耐熱性を予測するための機械学習モデルも構築した。
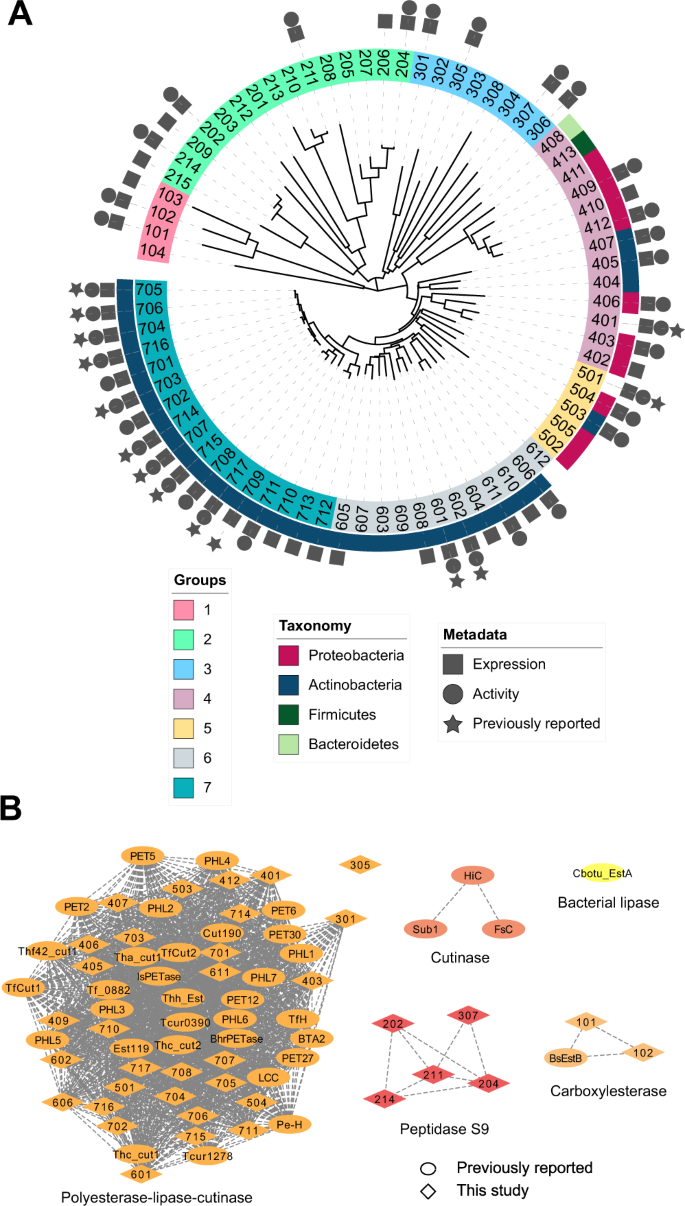
この2つの計算モデルを組み合わせることで、我戸らは未知の領域に踏み込むことができるようになった。1時間足らずで、2億5000万個以上のタンパク質をスクリーニングし、有望な候補を絞り込むことができた。さらにテストしたところ、36個のタンパク質がPETを分解できることが確認され、そのうち24個はこれまで科学文献に記載されていなかったものであった。
重要なのは、そのうちの数種類が非晶質PETよりも結晶質PETの分解に優れていたことです。
研究チームは、機械学習法によって抽出された酵素の中には、結晶性PETと非晶質PETを同じように分解できるものがあることに着目した。これらの酵素は、プラスチックの結合を軟化させるための前処理を必要としなかったのである。
「前処理を省くことで、この技術は、石油を使ったバージンPETの生産よりも実際に安価な工業規模のPETリサイクルを可能にします」と、Beckhamは付け加えました。「さらに良いことに、関連するエネルギーと温室効果ガスの排出を削減することができます。
<関連情報>
- https://www.nrel.gov/news/features/2022/scientists-discover-enzymes-cheaper-to-recycle-waste-polyester-textiles-and-bottles-than-making-from-petroleum.html
- https://www.nature.com/articles/s41467-022-35237-x
耐熱性ポリ(エチレンテレフタレート)ヒドロラーゼの足場を自然の多様性から調達すること Sourcing thermotolerant poly(ethylene terephthalate) hydrolase scaffolds from natural diversity
Erika Erickson,Japheth E. Gado,Luisana Avilán,Felicia Bratti,Richard K. Brizendine,Paul A. Cox,Raj Gill,Rosie Graham,Dong-Jin Kim,Gerhard König,William E. Michener,Saroj Poudel,Kelsey J. Ramirez,Thomas J. Shakespeare,Michael Zahn,Eric S. Boyd,Christina M. Payne,Jennifer L. DuBois,Andrew R. Pickford,Gregg T. Beckham & John E. McGeehan
Nature Communications Published:21 December 2022
DOI:https://doi.org/10.1038/s41467-022-35237-x
Abstract
Enzymatic deconstruction of poly(ethylene terephthalate) (PET) is under intense investigation, given the ability of hydrolase enzymes to depolymerize PET to its constituent monomers near the polymer glass transition temperature. To date, reported PET hydrolases have been sourced from a relatively narrow sequence space. Here, we identify additional PET-active biocatalysts from natural diversity by using bioinformatics and machine learning to mine 74 putative thermotolerant PET hydrolases. We successfully express, purify, and assay 51 enzymes from seven distinct phylogenetic groups; observing PET hydrolysis activity on amorphous PET film from 37 enzymes in reactions spanning pH from 4.5–9.0 and temperatures from 30–70 °C. We conduct PET hydrolysis time-course reactions with the best-performing enzymes, where we observe differences in substrate selectivity as function of PET morphology. We employed X-ray crystallography and AlphaFold to examine the enzyme architectures of all 74 candidates, revealing protein folds and accessory domains not previously associated with PET deconstruction. Overall, this study expands the number and diversity of thermotolerant scaffolds for enzymatic PET deconstruction.