生体膜内で機能しているたんぱく質の真の姿の解析が可能に
2019-09-06 科学技術振興機構
ポイント
- 従来の単粒子解析法は目標分子を取り出す必要があり、高分子が生体膜内に存在する状態(実環境下)での構造解析は困難だった。
- 実環境下での構造解析を可能とするIBSA(Image Based Structure Analysis)法を実現した。
- 本開発によって、生体膜内でのたんぱく質の構造解析が可能となり、生体機能の解明や創薬研究でクライオ電子顕微鏡が大いに貢献できると期待される。
JST(理事長 濵口 道成)は、産学共同実用化開発事業(NexTEP)の開発課題「実環境高分解能3次元生体構造解析システム」の開発結果を成功と認定しました。
この開発課題は、東京医科歯科大学の藤吉 好則 特別栄誉教授(開発当時 名古屋大学 特任教授)らの研究成果を基に、平成27年3月から平成31年3月にかけて日本電子株式会社(代表取締役社長 兼 COO 大井 泉、本社住所 東京都昭島市武蔵野3-1-2、資本金100億3,774万円)に委託して、同社EM事業ユニットにて企業化開発を進めていたものです。
受容体やチャネルなどの生体内高分子の3次元構造解析において、従来の単粒子解析法注1)ではたんぱく質の精製が必要であり、実際に生体膜内で機能しているたんぱく質の真の姿を解析することは困難でした。そのため生体内高分子の構造と機能の相関の研究には、生体膜中に存在する状態で立体構造を分子レベルで解明できる手法が求められていました。
この問題を解決したのが、藤吉教授らが開発したIBSA(Image Based Structure Analysis)法です。IBSA(イブサ)法は、単粒子解析法の長所を生かしながら解析結果の信頼性を上げ、生体膜中のたんぱく質の構造を解析する手法です。
本開発では、日本電子の最新鋭クライオ電子顕微鏡にIBSA法を用いてたんぱく質の3次元構造解析を行い、0.3ナノメートル(nm)以下の解像度でその構造を得ることができました。
今後、IBSA法に適した試料作製方法や粒子の抽出方法など知見を蓄積することで、生体内の膜たんぱく質の構造がより高解像度で得られるようになり、生体機能の解明や創薬研究で大いに貢献できると期待されます。
産学共同実用化開発事業(NexTEP)は、大学等の研究成果に基づくシーズを用い、民間企業が単独で事業化することが困難で、開発リスクが高く規模の大きい開発を支援し、実用化を後押しする事業です。
詳細情報 https://www.jst.go.jp/jitsuyoka/
<背景>
クライオ電子顕微鏡法の1つである単粒子解析法は、X線結晶構造解析に必須な試料の結晶化が必要なく、また、核磁気共鳴分光法注2)のように試料の分子量の上限がないという特徴があります。さらに、ごくわずかな水溶液試料で原子レベルの分解能を達成できるため、構造解析手法の基盤技術の1つとしてその立場を確立しています。しかしこの手法は、ランダムに配列された目標分子の画像を多数使用します。そのため目標分子を細胞内から取り出し、精製する操作が必要になり、生体内に存在する状態を捉えることはできませんでした。特に細胞膜中に存在するたんぱく質は、界面活性剤で膜から剥がす際に分子構造が変化してしまうことなどにより、細胞膜に入り込んだ状態での機能の解明が困難な場合があり、構造と機能の相関の研究には、細胞膜中に存在する状態で立体構造を分子レベルで解明できる手法が求められていました。
この問題を解決したのが、東京医科歯科大学の藤吉 好則 特別栄誉教授(開発当時 名古屋大学 特任教授)らが開発したIBSA(Image Based Structure Analysis)法です。IBSA法は、冷凍状態でステージ上に固定された試料の傾斜像と非傾斜像を撮影することで、単粒子解析法の長所を生かしながら3次元再構成の信頼性を向上させ、生体膜内で機能しているたんぱく質の構造を解析する手法です。
<開発内容>
本開発では、日本電子株式会社が市販する原子分解能電子顕微鏡に試料の自動搬送や液体窒素の自動供給機構を持つクライオステージを組み込むことでクライオ電子顕微鏡を完成させました(図1)。IBSA法についてはアルゴリズムの構築、検証およびIBSA法に適した試料作製条件の検討などを行いました。クライオ電子顕微鏡を用いた構造解析を行った結果、酵素の1つであるβ-galactosidase注3)について、FSC(Fourier Shell Correlation)法注4)による評価(FSC=0.143)で分解能0.27nmを得ることができました(図2)。
<期待される効果>
クライオ電子顕微鏡での構造解析は、X線構造解析と異なり、試料が結晶構造である必要がないため、結晶を作製することができないたんぱく質の構造解析が可能です。さらに、クライオ電子顕微鏡にIBSA法を適用することで、実際に生体膜内で機能しているたんぱく質の構造解析も可能になり、生体機能の解明や創薬研究で大いに貢献できると期待されます。
<参考図>

図1 クライオ電子顕微鏡
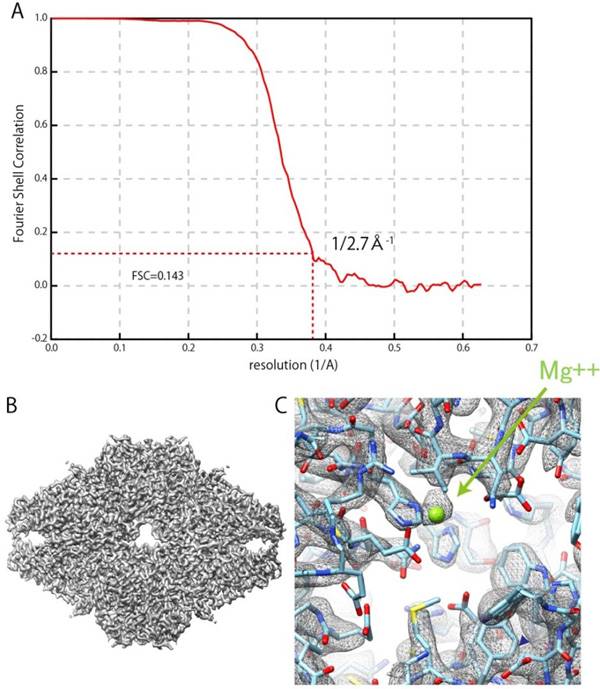
図2 β-galactosidaseの3次元再構成結果
(A)FSCプロット。赤破線はFSC=0.143を示しており、赤実線と最初に交差した箇所から分解能0.27nmを定義している。
(B)3次元再構成したマップと原子モデル。
(C)酵素活性部位のMg2+イオン。
<用語解説>
- 注1)単粒子解析法
- 電子顕微鏡において分離された多数の単一粒子を観察し、画像処理によって粒子の詳細な構造を得る手法で、X線結晶構造解析で必須な試料の結晶化が不要である。
- 注2)核磁気共鳴分光法
- 強い磁場中に試料を置き、核スピンの向きを揃えた分子にパルス状のラジオ波を照射し、核磁気共鳴させた後、分子が元の安定状態に戻る際に発生する信号から分子構造を解析する手法。
- 注3)β-galactosidase
- 大腸菌由来のたんぱく質であり、四量体で機能し、ラクトースをグルコースとガラクトースに分解する酵素。X線構造解析により古くから構造解析されている。
- 注4)FSC(Fourier Shell Correlation)法
- 分解能推定法の1つ。電子顕微鏡写真から解析のために選び出した粒子をランダムに2つのグループにあらかじめ分け、それぞれのグループで独立に3次元構造を再構成する。得られた独立の3次元構造をフーリエ変換し、任意の分解能の範囲ごとに2つのグループ間の相関を計算する。相関値であるFSCが0.143に初めて到達した分解能位置を解析結果の分解能として定義する場合が多い。
<お問い合わせ先>
<開発内容に関すること>
石川 勇(イシカワ イサム)
日本電子株式会社 EM事業ユニット
<JST事業に関すること>
沖代 美保(オキシロ ミホ)
科学技術振興機構 産学共同開発部