2023-12-22 東京大学,理化学研究所
発表のポイント
- 顕微鏡やDNAシークエンサーから生まれる大量の分子・細胞計測データから、特定の対象を識別するための微小な識別子マテリアルを、計算論的デザインを通じて実現しました。
- 実データのシミュレーションを経由するReal2Sim2Real機械学習により、ランダムに生成した微小な識別子を自在に読み出せるようにする新たな識別子設計法を提案し、実際に識別子を作り実証しました。
- この微小識別子マテリアルを用いて生成される個々の細胞や分子に紐づいた大量の多角的解析データは、今後の進展が極めて重要となるマルチモーダルAIの開発を始め、基礎研究から医薬工学応用まで、幅広い活用が期待されます。
計算科学の手法で識別子を作って読み出す
発表概要
東京大学先端科学技術研究センターの太田禎生准教授と油谷浩幸シニアリサーチフェロー、理化学研究所革新知能統合研究センターの河﨑史子研究員(研究当時)、三森隆広研究員(研究当時)は、東京大学大学院情報理工学系研究科の佐藤一誠教授、ブリティッシュコロンビア大学の谷内江望教授と共同で、細胞スケールで使える多種多様な光学識別子を計算論的手法でデザインする手法を開発しました。
微小スケールの識別子は、微小な世界の計測情報を時間や装置を越えて束ねる用途において、様々な活用が期待されてきました。しかし、大きな世界においてルールに従って設計したバーコードやQRコードをプリントするようにはいかず、微小で精密な識別子を、大量かつ実用的に作製するのは困難でした。そこで本研究では「ボトムアップでランダムな生成手法により、大量の異なるパターンを持った微小スケールの識別子を発生させ、あらかじめシミュレーションデータを用いて訓練しておいた機械学習モデルにより自在にデコード(注1)できるようにする」という逆転のアプローチに着想し、これを実験的に実現しました。
そしてこの識別子を、顕微鏡とゲノムシークエンシングという異なる2つの計測で識別するための実験的デザインを実現し、実際に多数の1細胞から取得した顕微鏡画像データと遺伝子配列データをつなぐ識別子として実証しました。この識別子と機械学習手法は、大量の1細胞や分子を混在させたままで経時的にトラッキングしたり、装置を越えて多角的に観測したりするためのツールの開発に役立つと期待されます。
本研究成果は、国際雑誌「Advanced Optical Materials」(2023年12月20日)にオンライン掲載されました。
ー研究者からのひとことー
本研究は、生命計測のための小さな「光記憶媒体ゲル」という新コンセプトを、初めて実証したものです。光学、化学、情報科学、マテリアル工学まで異分野の研究者が協力して、楽しく知的な挑戦を続け、苦難を乗り越えて実現したコンセプトでもあります。支援して下さった機関と皆様に心から感謝します。本技術は、細胞・分子データの統合を目指して日々進化しており、AI開発、光学解析、生命医科学分野の進歩への貢献を目指します。(太田禎生准教授)
発表内容
バーコードやQRコードは光で見える情報を用いて、特定の対象を識別・追跡できる、物流などに欠かせない識別子です。しかし細胞や分子など、微小なスケールの物体追跡のためにこのような識別子を精緻かつ大量に作り出すことは困難でした。本研究では、微小なスケールで使える大量の異なる光学パターンを持った微小識別子を、計算論的手法でデザインする手法を開発しました。この手法は、「ボトムアップでランダムな生成手法により、大量の異なるパターンを持った微小スケールの識別子を発生させ、あらかじめシミュレーションデータを用いて訓練しておいた機械学習モデルにより自在にデコードする」という逆転のアプローチに基づいています。
より具体的には、色の異なる微小なビーズを数十種類、数億個用意し、このビーズの多様な組み合わせを光学識別子として用いました。本手法では、個々の微小ビーズの顕微鏡画像からシミュレーションで合成した、多様なビーズの組み合わせ画像を用いてニューラルネットワークによるデコーダーを訓練しました。そして実験的に合成した光学識別子に対して、この訓練したネットワークを適用し、正確にデコードできることを確認しました(図1)。
この技術の実証例として本研究では、微小スケールで流体の挙動を制御するマイクロ流体技術と呼ばれる方法を用いて実物の微小ビーズを用いた光学識別子を作製し、細胞に付加しました。そして光学識別子をデコードすることで、顕微鏡イメージ中の個々の細胞が識別できることを示しました(図1赤矢印)。この研究は、(1)現実的には作製不可能な大量のラベル付き教師データをシミュレーションで大量合成してデコーダーを構築し、(2)構築したデコーダーを実物の光学識別子の解析に応用した「Real2Sim2Real(注2)」な識別子設計アプローチです。
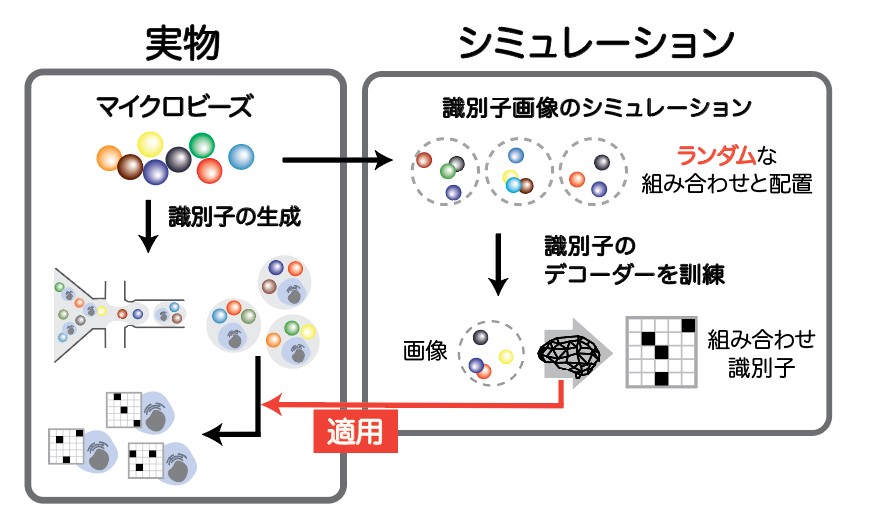
図1:本研究のコンセプト
さらに本研究では、ケミカルバイオロジーの手法を駆使して、光学識別子が持つ識別情報をシーケンシングと呼ばれる分子計測装置でも読み取れるようにしました。具体的には、微小ビーズの蛍光シグナルに対応した配列を持つDNA分子をビーズに結合し、顕微鏡観察では蛍光ビーズの組み合わせパターンを、分子計測(シーケンシング)ではDNA配列の組み合わせパターンを読み出しました(図2)。この新たな識別子を用いた細胞識別法、Image-DNA dual barcoding(ID-coding)(注3)は、顕微鏡とゲノムシークエンシングという異なる2つの計測で識別するための実験的デザインを実現し、実際に多数の1細胞から取得した顕微鏡画像データと遺伝子配列データをつなぐ識別子として実証しました。この識別子と機械学習手法は、大量の1細胞や分子を混在させたままで経時的にトラッキングしたり、装置を越えて多角的に観測したりするためのツールの開発に役立ちます。このような1細胞や分子の多角計測技術は、人体で起こる生物学的現象の理解に役立ち、今後の進展が重要となるマルチモーダルAI(注4)の開発に重宝されていくと期待されます。また今後その実用性を、疾患関連サンプルを含む様々なサンプルで実証していくことが期待されます。
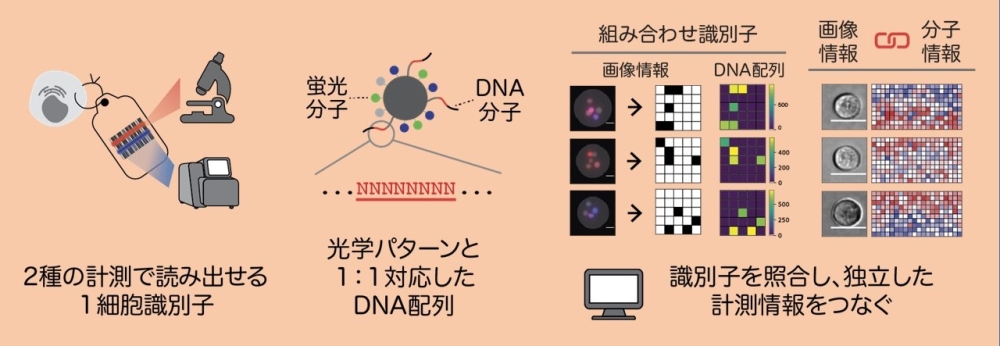
図2:識別子デザイン法を応用したImage-DNA dual barcoding
発表者・研究者等情報
東京大学
先端科学技術研究センター
太田 禎生(准教授)
油谷 浩幸(シニアリサーチフェロー)
大学院情報理工学系研究科
佐藤 一誠(教授)
理化学研究所
革新知能統合研究センター
河﨑 史子(研究当時:研究員)
現:同客員研究員、東京大学定量生命科学研究所 助教
三森 隆広(研究当時:研究員)
現:同客員研究員、早稲田大学理工学術院総合研究所 次席研究員
森 有加(研究当時:技官)
現:東京大学医科学研究所附属病院 学術専門職員
ブリティッシュコロンビア大学
谷内江 望(教授)
兼:先端科学技術研究センター 客員教授
論文情報
- 雑誌:Advanced Optical Materials(12月20日)
- 題名:Computational Design of Synthetic Optical Barcodes in Microdroplets
- 著者:Fumiko Kawasaki, Takahiro Mimori, Yuka Mori, Hiroyuki Aburatani, Nozomu Yachie, Issei Sato*, Sadao Ota*
*責任著者 - DOI:10.1002/adom.202302564
研究助成
本研究は、科学技術振興機構(JST)CREST「多次元・ネットワーク化計測による細胞外微粒子の多様性と動態の解明(課題番号:JPMJCR19H1)」、科研費「ゲノム編集iPS細胞を用いた超網羅的表現型解析法の開発(課題番号:JP21H04636)、「接着シンギュラリティ細胞の動態から分子まで捉えるネットワーク化計測(課題番号:JP21H00416)」などの支援により実施されました。
用語解説
(注1)デコード
本研究においては、顕微鏡画像から識別子を特定すること。
(注2)Real2Sim2Real
仮想環境で開発したアルゴリズムを実世界に適合させる過程やそのための技術はSim2Realと呼ばれる。本研究では、実空間で取得したデータを起点としてシミュレーションと学習を行う“Real2Sim2Real”な手法を開発し、ランダムな識別子のデコードを実現した。
(注3)Image-DNA dual barcoding(ID-coding)
本研究で開発したものであり、画像情報とDNA配列という異なる2つの計測で読み出せる識別子を付与する方法を指す。
(注4)マルチモーダルAI
実世界に存在する対象の異なる側面を反映したマルチモーダル情報を認識し、対象の統合的な把握や、不完全な計測情報から全体像の復元を可能とするAI技術を指す。顕微鏡画像・DNAシークエンシングデータなど異種モダリティの同時計測技術により、細胞情報をより多角的に捉えるマルチモーダルAI技術の発展が見込まれる。
問合せ先
東京大学 先端科学技術研究センター ロボティック生命光学分野
准教授 太田 禎生(おおた さだお)