2019-12-06 理化学研究所,微生物化学研究所
理化学研究所(理研)環境資源科学研究センターケミカルバイオロジー研究グループの永澤生久子基礎科学特別研究員、室井誠専任研究員、川谷誠専任研究員、長田裕之グループディレクターと微生物化学研究所第1生物活性研究部の川田学部長らの共同研究チームは、熱安定性の変化を見ることで化合物と結合するタンパク質(標的タンパク質)を探索する手法を構築し、この手法を用いて、抗がん活性を持つ化合物の標的タンパク質の候補を同定することに成功しました。
本研究成果は、表現型スクリーニング[1]から見いだされた生理活性化合物の標的タンパク質の同定とその作用メカニズムの解明に貢献すると期待できます。
今回、共同研究チームは、二次元電気泳動[2]を用いたプロテオーム解析[3]により、化合物との結合によって熱安定性が変化するタンパク質を網羅的に解析する手法「2DE-CETSA」を構築し、ヒトがん細胞株に対して細胞増殖阻害作用を持つ化合物NPD10084の標的タンパク質の候補として、解糖系[4]の代謝酵素の一つであるPKM2[5]を同定しました。
本研究は、米国の科学雑誌『Cell Chemical Biology』の掲載に先立ち、オンライン版(12月5日付:日本時間12月6日)に掲載されます。
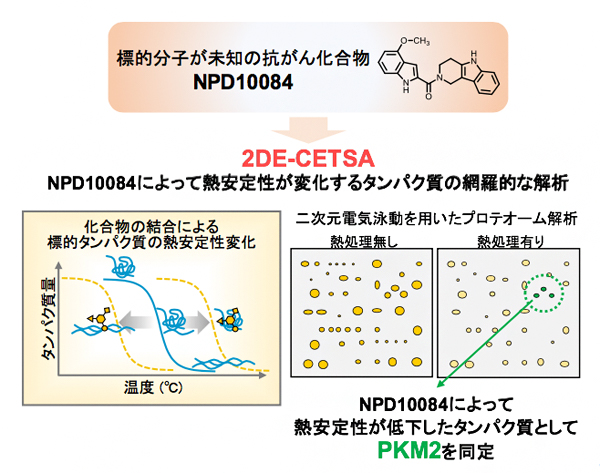
図 「2DE-CETSA」によるNPD10084の標的タンパク質の解析
背景
表現型スクリーニングにより見いだされた生理活性化合物の標的分子の解明は、創薬研究において重要なステップですが、共通の正攻法がないため多くの時間と労力を要します。理研環境資源科学研究センターのケミカルバイオロジー研究グループではこれまでに、化合物ビーズを用いたプルダウン法[6]や、細胞形態やプロテオーム変化をもとに細胞応答を解析する手法を独自に開発してきました注1-3)。本研究では、これまでとは異なる原理の解析法を開発することで、標的分子同定のさらなる迅速化と高精度化を目指しました。
細胞サーマルシフトアッセイ(CEllular Thermal Shift Assay;CETSA)は、化合物の結合によるタンパク質の熱安定性変化を検出することで標的分子を同定する手法であり、化合物の修飾を必要とせず、煩雑な操作を伴わない点で優れています。しかしこれまで、標的タンパク質が未知の場合にはCETSAは適応できませんでした。
そこで共同研究チームは、二次元電気泳動によるプロテオーム解析を用いて、化合物が熱安定性を変化させるタンパク質を網羅的に探索する方法の開発を試みました。
注1)Kanoh, N. et al., Photo-cross-linked small-molecule affinity matrix for facilitating forward and reverse chemical genetics, Angew Chem Int Ed Engl, 44, 3559-3562, 2005.
注2)Muroi, M. et al., Application of proteomic profiling based on 2D-DIGE for classification of compounds according to the mechanism of action, Chem Biol, 17, 460-470, 2010.
注3)Futamura, Y. et al., Morphobase, an encyclopedic cell morphology database, and its use for drug target identification, Chem Biol, 19, 1620-1630, 2012.
研究手法と成果
共同研究チームは、化合物が熱安定性を変化させるタンパク質を網羅的に探索するシステム「2 Dimensional gel Electrophoresis – CETSA(2DE-CETSA)」を構築しました。2DE-CETSAの手順として、化合物を添加したがん細胞の抽出液を熱処理し、遠心分離後の可溶性画分を二次元電気泳動により展開します。その後、溶媒のみを添加したコントロール条件と比較することで、化合物処理によって熱安定性が変化したタンパク質を見いだすことができます(図1)。
最初に、構築した2DE-CETSAを評価するため、モデル化合物として抗生物質の一種であるゲルダナマイシンを用いた解析を実施したところ、ゲルダナマイシンの標的分子であるHSP90[7]タンパク質の熱安定性の上昇を見いだすことに成功しました。
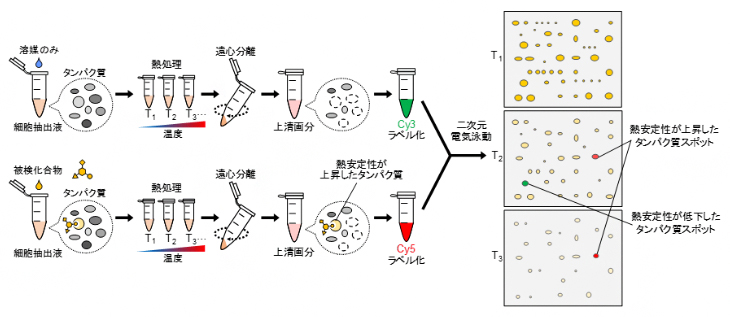
図1 2DE-CETSAの手順
細胞抽出液に標的分子が未知の被検化合物を添加し、熱処理後、蛍光色素Cy5でラベル化した可溶性画分を二次元電気泳動により展開する(下段)。上段の化合物非添加条件(蛍光色素Cy3でラベル化)と比較して、化合物の処理によって熱安定性が変化したタンパク質が、赤または緑のスポットとして検出される。
次に、2DE-CETSAを用いて、作用機序が未知の化合物NPD10084の標的タンパク質の同定を試みました。NPD10084は、理研NPDepo化合物ライブラリー[8]から大腸がん細胞株に対する細胞増殖阻害作用を指標に見いだした化合物です。2DE-CETSA解析により、NPD10084処理によって熱安定性が低下するタンパク質として、解糖系の代謝酵素の一つであるPKM2を同定しました(図2)。

図2 NPD10084によるPKM2タンパク質の熱安定性の低下
上:NPD10084処理によって熱安定性が低下したタンパク質のスポット(緑色)が五つ検出され、これらのスポットは全てPKM2であることが明らかとなった。
下:NPD10084処理によってPKM2タンパク質の熱安定性が低下することを、ウェスタンブロット(抗体を用いて特定のタンパク質の存在を検出する手法)により確認した。
検証を進めると、NPD10084はPKM2のピルビン酸キナーゼ[4]活性には影響を与えない一方で、PKM2と細胞間接着と遺伝子の転写制御に関与するβ-カテニン[9]およびシグナル伝達[10]を担う転写因子[11]STAT3[12]とのタンパク質間相互作用を阻害し、下流のシグナル伝達を抑制することを見いだしました。さらに、NPD10084はヒト大腸がん細胞株を用いたマウスゼノグラフトモデル[13]において、腫瘍の増殖を抑制し、腫瘍内においてもPKM2の下流シグナル伝達を阻害することを明らかにしました(図3)。
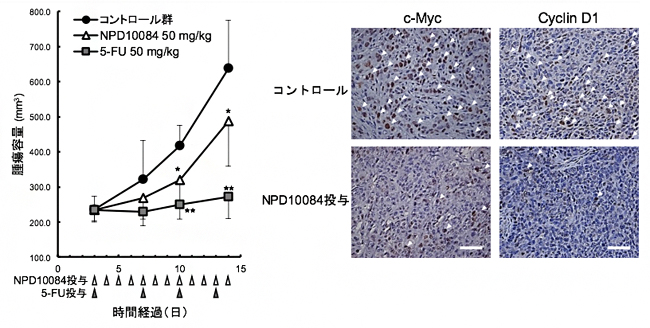
図3 NPD10084による腫瘍増殖抑制(左)と腫瘍内におけるシグナル伝達阻害
左:ヒト大腸がん細胞株HCT116を用いたマウスゼノグラフトモデルにおいて、NPD10084(△)またはフルオロウラシル(5-FU、ポジティブコントロール、□)を投与し、腫瘍容量を計測した結果、コントロール群(●)に比較して、NPD10084投与群では腫瘍の増殖が抑制された。
右:NPD10084を投与したマウスの腫瘍では、PKM2の下流で調節されるc-Myc(細胞の増殖分化などに関わる転写因子)およびCyclin D1(細胞周期の制御に関わるタンパク質)の発現量が低下した。スケールバーは50μm。
今後の期待
創薬研究において表現型スクリーニングは革新的新薬の発見に有効な方法として利用されていますが、スクリーニングから見いだされた生理活性化合物の標的分子を明らかにすることは容易ではありません。本研究で構築した2DE-CETSAは、この標的同定の迅速化に役立ち、新しい治療薬の開発を促進させると期待できます。
実際に本研究では、2DE-CETSAにより抗がん活性を持つ化合物NPD10084の標的タンパク質の候補としてPKM2を同定し、既存の化合物とは異なるユニークな作用機序を明らかにしました。このような新しい活性を持つ抗がん化合物の発見は、がん治療法のさらなる発展に重要と考えられます。
補足説明
1.表現型スクリーニング
細胞や臓器などの生命現象に影響を与える作用を指標として、新しい化合物を探索する手法。
2.二次元電気泳動
タンパク質を二段階の電気泳動により二次元に分離する手法。通常は、一次元目は等電点電気泳動により分離し、二次元目はSDS-PAGEにより分子量で分離する。
3.プロテオーム解析
生物の持つタンパク質の構造や機能、発現量を網羅的に解析する手法。
4.解糖系、ピルビン酸キナーゼ
解糖系とは、グルコースをピルビン酸や乳酸に異化する代謝経路。ピルビン酸キナーゼは、解糖系においてホスホエノールピルビン酸とADPからピルビン酸とATPを生成する。
5.PKM2
ピルビン酸キナーゼアイソザイムM2。解糖系においてホスホエノールピルビン酸とADPとから、ピルビン酸とATPを合成する酵素。
6.化合物ビーズを用いたプルダウン法
化合物を固定化したビーズを用いて、細胞抽出液から化合物に結合するタンパク質を探索する手法。
7.HSP90
ヒートショックプロテイン(HSP)ファミリーの一種。タンパク質のフォールディングを制御する分子シャペロンとして機能する。
8.理研NPDepo化合物ライブラリー
理研が保有する天然化合物・合成化合物のライブラリー。
9.β-カテニン
細胞間接着と遺伝子の転写制御に関与するタンパク質。一部のがんではβ-カテニンの持続的な蓄積により下流の標的遺伝子の発現が亢進することが知られている。
10.シグナル伝達
細胞において、ある種の情報(シグナル)が他の種類の情報に変換されていく過程。細胞内の因子が次々と情報を受け渡し、最終的に細胞の機能変化や遺伝子の発現調節などを起こす。
11.転写因子
遺伝子の発現を調節するタンパク質。DNA上に存在する遺伝子の発現を制御する領域(エンハンサー、プロモーター、サイレンサーなど)に結合し、DNAを鋳型としてRNAが産生(転写)される時期や量を調節する。
12.STAT3
シグナル伝達を担うSTATファミリー転写因子の一種。チロシンキナーゼによるリン酸化を受け核内に移行し、標的遺伝子の転写を活性化する。
13.マウスゼノグラフトモデル
ヒト由来のがん細胞・組織を免疫不全マウスに移植した実験モデル。
共同研究チーム
理化学研究所 環境資源科学研究センター ケミカルバイオロジー研究グループ
基礎科学特別研究員 永澤 生久子(ながさわ いくこ)
専任研究員 室井 誠(むろい まこと)
専任研究員 川谷 誠(かわたに まこと)
グループディレクター 長田 裕之(おさだ ひろゆき)
微生物化学研究所 第1生物活性研究部
部長 川田 学(かわだ まなぶ)
主任研究員 大石 智一(おおいし ともかづ)
研究員 大庭 俊一(おおば しゅんいち)
研究支援
本研究の一部は、日本学術振興会(JSPS)科学研究費補助金・新学術領域研究(研究領域提案型)「化学コミュニケーションのフロンティア(領域代表:掛谷秀昭)」「プロテオミクスをベースにした化学シグナル解析技術開発(研究代表者:長田裕之)」、同新学術領域研究(研究領域提案型)「学術研究支援基盤形成(研究支援代表者:井上純一郎)」「先端モデル動物支援プラットフォーム(AdAMS)(研究支援分担者:長田裕之)」、同基盤研究C「プロテオーム解析を用いたパスウェイ解析に基づく化合物評価システムの開発(研究代表者:室井誠)」、日本医療研究開発機構(AMED)次世代がん医療創生研究事業「ケミカルバイオロジーを基盤としたがん代謝制御薬剤の開発(研究代表者:長田裕之)」による支援を受けて行われました。
原論文情報
Ikuko Nagasawa, Makoto Muroi, Makoto Kawatani, Tomokazu Ohishi, Shun-ichi Ohba, Manabu Kawada, Hiroyuki Osada, “Identification of a small compound targeting PKM2-regulated signaling using 2D gel electrophoresis-based proteome-wide CETSA”, Cell Chemical Biology, 10.1016/j.chembiol.2019.11.010![]()
発表者
理化学研究所
環境資源科学研究センター ケミカルバイオロジー研究グループ
基礎科学特別研究員 永澤 生久子(ながさわ いくこ)
専任研究員 室井 誠(むろい まこと)
専任研究員 川谷 誠(かわたに まこと)
グループディレクター 長田 裕之(おさだ ひろゆき)
微生物化学研究所 第1生物活性研究部
部長 川田 学(かわだ まなぶ)
報道担当
理化学研究所 広報室 報道担当
公益財団法人微生物化学研究会
微生物化学研究所(微化研)知的財産情報室 室長 山﨑勝久