2019-09-19 名古屋大学,国立遺伝学研究所,科学技術振興機構
ポイント
- 光学顕微鏡より小さいものを見ることができる超解像蛍光顕微鏡を駆使して、生きた細胞のゲノムDNAを観察し、その動きを追跡して統計的に分析しました。
- ゲノムDNAは規則正しい構造の中で振動しているのではなく、液体のように不規則に流動的に動いていることを明らかにしました。
- ゲノムDNAの中で速く動く部分と遅く動く部分があり、細胞の核内にそれぞれ特徴的な塊をつくるとともに、その比率が細胞の状態に応じて大きく変化することを発見しました。
名古屋大学 大学院工学研究科のS.S.Ashwin 特任助教、笹井 理生 教授のグループは、国立遺伝学研究所の野崎 慎 日本学術振興会特別研究員、前島 一博 教授のグループとの共同研究により、ヒト細胞核の中でゲノムDNAが多様で流動的な動きを示すことを明らかにしました。共同研究グループは超解像蛍光顕微鏡注1)を駆使して、生きた細胞内のゲノムDNAを観測し、その動きを統計的に分析しました。その結果、ゲノムDNAの中で速く動く部分と遅く動く部分があり、それぞれ特徴的な塊を形成して、細胞の状態に応じてその比率が大きく変わることを発見しました。
本研究によって、DNAは生きた細胞の中で大きく揺らいでいて、その動きと細胞の性質に密接な関連があることが明らかになりました。この成果は、DNAの変化に伴って生じる細胞の異常や関連する疾患の理解につながることが期待されます。
この研究成果は、2019年9月16日付米国科学アカデミー紀要「Proceedings of the ational Academy of Sciences of the United States of America」オンライン版に掲載されました。
本研究は、科学技術振興機構 戦略的創造研究推進事業(CREST)(JPMJCR15G2)、科研費 新学術領域研究「クロマチン潜在能」(JP19H05258、JP19H05273)科研費 新学術領域研究「先進ゲノム支援」(JP16H0627)、科研費基盤研究B(JP19H01860、JP16H04746)、武田科学振興財団、RIKEN Pioneering Project、遺伝研共同研究(2016-A2(6))の支援を受けました。
<研究背景と内容>
私たちの体の設計図であるゲノムDNAは、全長約2メートルに及ぶひも状の物質ですが、このゲノムDNAは10万分の1メートルほどの小さな細胞核の中に収納されています。つまり、全長200キロメートルの鉄道線路を折りたたんで1メートルの机の上に置くのと同じ比率で圧縮されているのです。しかしながら、この圧縮がどのように実現され、細胞核の中にゲノムDNAがどのように収納されているのか、その様子は、長い間、解明されていませんでした。
DNAは「ヒストン」という樽状のたんぱく質に巻かれることで「ヌクレオソーム」を作っており(図1上段)、ヌクレオソームとこれに結合する他のたんぱく質を合わせて「クロマチン」と呼んでいます。これまで教科書に書かれていた定説によれば、このクロマチンは、らせん状に規則正しく折りたたまれ、さらに巻かれて階層構造を作ることにより、上記のような高度の圧縮を実現していると考えられてきました(図1中段左)。しかし、国立遺伝学研究所の前島教授らは、クライオ電子顕微鏡注2)やX線散乱注3)の実験に基づいて、細胞核内のクロマチンには規則正しい階層構造はほとんど存在せず、クロマチンは不規則に細胞核内に収納されているという新しい説を提唱してきました(図1中段右)。さらに2012年頃になって、前島教授のグループは1個1個のヌクレオソームを観察できる超解像蛍光顕微鏡による解析方法を開発して、ヌクレオソームの振る舞いを生きたままの細胞で観察することに成功しました(図2)。具体的には、細胞1個に3000万個程度存在するヌクレオソームをまばらに蛍光たんぱく質で目印(標識)を付けることにより(図2A、B)、個々のヌクレオソームの動きを正確に調べることが可能となったのです(図2C、動画)。
本研究では、こうして得られた個々のヌクレオソームの動きを追跡し、観測データからノイズの影響を減らす繰り返し計算法注4)を用いて統計的に分析することで、クロマチンは規則正しい構造をとって振動しているのではなく、液体のように不規則に流動的に動いていることを明らかにしました。そして、クロマチンの細胞核内での収納方法について、新しい説の優位性を明確に示しました。また、この流動的な動きを詳しく調べることにより、速く動くクロマチンと遅く動くクロマチンの集団があり(図3)、速いクロマチンと遅いクロマチンは細胞核の中でそれぞれ特徴的な塊(ドメイン領域)を作っていること、速いクロマチンと遅いクロマチンの比率やドメインの配置は細胞の状態によって変化することを発見しました。遅いクロマチンは、細胞核膜周辺のラミン注5)や核の中のコヒーシン注6)と呼ばれるたんぱく質などによって、ヌクレオソームの動きが束縛されて産み出されていると思われます。こうして、細胞核の中で大きく流動的に動くクロマチンと、その運動の多様性を明らかにしました。
<成果の意義>
細胞の活動の多くは、DNAから遺伝情報を読み取る転写から始まりますが、この読み取り過程はDNAの動きによって大きく影響を受けると考えられ、DNAの動きと細胞の性質の関係は大きな注目を集めています。本研究で明らかになった速いクロマチンと遅いクロマチンの集団の分布や比率と細胞の性質との関係を知ることは、DNAの動きを調べる新しい見方となると思われます。
本研究で用いられた超解像蛍光顕微鏡による1分子ヌクレオソーム観測とその統計的分析を組み合わせた方法は、DNAの動きを調べる新しい強力な方法として、その有効性が示されました。これらはDNAの動きと細胞の性質の関係を解明する有力な手掛かりです。DNAからの遺伝情報の読み取りに異常が生じると、細胞のガン化、細胞死、さらには細胞分化の異常など、さまざまな細胞障害が生じることが近年分かってきました。本研究の成果によって、このような細胞の異常や関連疾患の理解が進むことが期待されます。
<参考図>
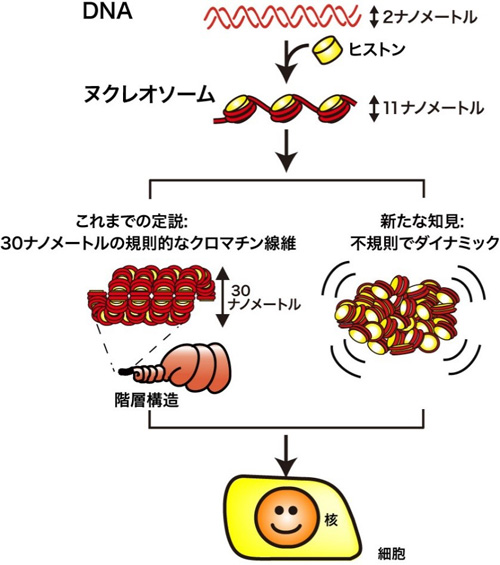
図1
(上段)DNAはヒストンに巻かれてヌクレオソームを形成する。ここでは3個のヌクレオソームが描かれているが、1つの細胞当たり、3000万個ものヌクレオソームが存在する。
(中段)DNAの細胞内収納に関するこれまでの定説(左)と現在の説(右)。
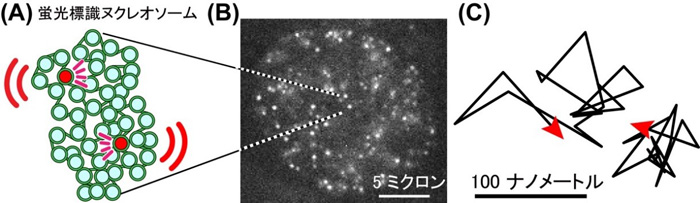
図2
(A)少数のヌクレオソームを蛍光標識する(赤)ことにより、ヌクレオソーム1個1個の動きを調べることができる。
(B)超解像蛍光顕微鏡による核内のヌクレオソーム画像。1個1個のドットが1個1個のヌクレオソームを示している。Nozaki et al.(2017)より転載。
(C)個々のヌクレオソームの動きを1コマ50ミリ秒で追跡した。
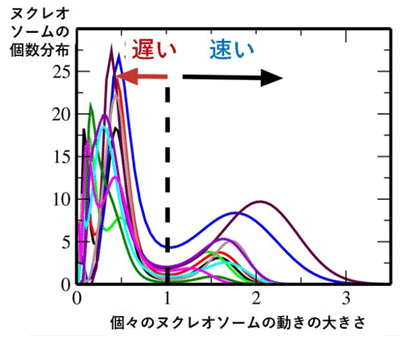
図3
観測された個々のヌクレオソームの動きの大きさを平均二乗変位注7)で測ると、ヌクレオソームの個数分布はふた山にわかれ、速いヌクレオソーム(クロマチン)と遅いヌクレオソーム(クロマチン)があることがわかる。図は10個の細胞についての観測結果を重ねて描いたもの。
(動画)https://youtu.be/rZuNIrxvdXk
超解像蛍光顕微鏡による細胞核内のヌクレオソームの動きの動画。1個1個のドットが1個1個のヌクレオソームを示している。1コマ50ミリ秒。
<用語解説>
- 注1)超解像蛍光顕微鏡
- 通常の光(可視光)を用いて顕微鏡観察する場合は、10の7乗分の1メートル程度の大きさのモノを解像するのが限界(光の回折限界)である。しかし、超解像蛍光顕微鏡はこの限界を超えて(超解像)、より小さな構造まで観察することができる。本研究では、10の8乗分の1メートル程度の大きさであるヌクレオソームをまばらに蛍光標識することで超解像を達成した。
- 注2)クライオ電子顕微鏡
- 生きた状態に近い生物材料を観察することができる電子顕微鏡。「生きている」状態を保存するために、細胞を急速凍結し、凍結した細胞を極低温下(-150度)で薄く切り(切片化)、その切片を極低温下でそのまま観察する。
- 注3)X線散乱
- たんぱく質などが集まった構造体にX線を当てると、その構造体の規則性に応じた散乱パターンが得られる。このことを利用し、構造を調べる方法。大型放射光施設SPring-8で強力なX線である放射光を用いて実験を行った。
- 注4)繰り返し計算法
- 劣化した画像データからノイズを減らして、鮮明な画像を回復する繰り返し計算法をヌクレオソームの動きを記録したデータに適用し、ノイズを減らして物理量の分布の特徴を抽出することを可能にした。
- 注5)ラミン
- 細胞核膜の繊維状たんぱく質で、核膜の裏側にメッシュ状に存在し、構造維持に働いていると考えられている。
- 注6)コヒーシン
- クロマチンを束ね、ループを作ることができるリング状のたんぱく質。もともとは2本の姉妹染色体を接着させるたんぱく質として発見された。
- 注7)平均二乗変位
- ある時間の間に粒子が移動した距離の二乗を平均した量。この量は、粒子が動く程度を表現するために使われる。
<論文タイトル>
- “Organization of fast and slow chromatin revealed by single-nucleosome dynamics”
(一分子ヌクレオソームの動きを調べることにより、速いクロマチンと遅いクロマチンの構成がわかる) - 著者名:S. S. Ashwin, Tadasu Nozaki, Kazuhiro Maeshima, and Masaki Sasai
- DOI:10.1073/pnas.1907342116
<お問い合わせ先>
<研究に関すること>
笹井 理生(ササイ マサキ)
名古屋大学 大学院工学研究科 教授
<JST事業に関すること>
川口 哲(カワグチ テツ)
科学技術振興機構戦略研究推進部
<報道担当>
名古屋大学 総務部 総務課 広報室
国立遺伝学研究所 リサーチ・アドミニストレーター室 広報チーム
科学技術振興機構 広報課



