2019-04-26 理化学研究所,日本医療研究開発機構
理化学研究所(理研)科技ハブ産連本部バトンゾーン研究推進プログラム横山特別研究室の横山茂之特別招聘研究員、生命機能科学研究センター非天然型アミノ酸技術研究チームの柳沢達男上級研究員(研究当時)らの共同研究グループは、様々な非天然型アミノ酸(人工アミノ酸)をタンパク質に導入できる「AF変異ピロリシルtRNA合成酵素(AF変異PylRS)[1]」と、14種類の人工アミノ酸との複合体の立体構造を網羅的に解析し、AF変異PylRSがこれまでの酵素学の常識を覆す能力を持つ酵素であることを明らかにしました。
本研究成果により、AF変異PylRSと人工アミノ酸複合体の立体構造をもとにした新しい人工アミノ酸の合理的な設計が可能になります。設計した人工アミノ酸を利用して作製された高機能タンパク質は、医療や産業へ応用されると期待できます。 現在、AF変異PylRSを用いて多様な人工アミノ酸が導入されたタンパク質は生命現象の解明や抗体医薬などさまざまな用途に利用されています。しかし、AF変異PylRSが多様なアミノ酸を取り込むメカニズムについては、全く分かっていませんでした。
今回、共同研究グループはX線結晶構造解析[2]により、さまざまな官能基を持つ14種類の人工アミノ酸がどのようにAF変異PylRSに結合しているかを詳しく調べました。その結果、AF変異PylRSは基質[3]であるそれぞれの人工アミノ酸に特異的に対応し、異なる様式でそれぞれを認識していることが分かりました。通常、多種類の基質を認識する酵素は基質間での共通部分を認識することから、AF変異PylRSは、従来の酵素学の常識に反する全く新しい仕組みを持つといえます。
本研究は、米国の科学雑誌『Cell Chemical Biology』オンライン版(4月25日付け、日本時間4月26日)に掲載されます。
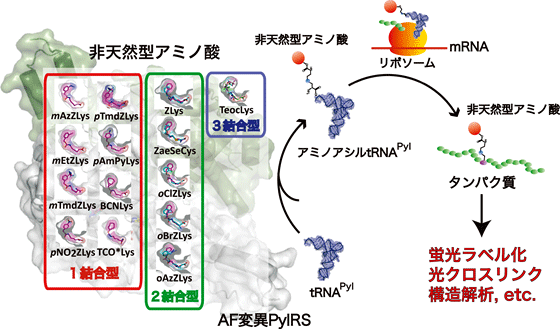
図 AF変異PylRSによる人工アミノ酸のタンパク質への導入およびその応用
※共同研究グループ
- 理化学研究所
- 科技ハブ産連本部バトンゾーン研究推進プログラム 横山特別研究室
特別招聘研究員 横山 茂之(よこやま しげゆき)
研究員(研究当時) 倉谷 光央(くらたに みつお)
研究員 関 英子(せき えいこ)
- 生命機能科学研究センター 非天然型アミノ酸技術研究チーム チームリーダー 坂本 健作(さかもと けんさく)上級研究員(研究当時)柳沢 達男(やなぎさわ たつお)
大阪大学大学院 薬学研究科 創成薬学専攻
- 助教 樋野 展正 (ひの のぶまさ)
※研究支援
本研究は、日本医療研究開発機構(AMED)創薬等先端技術支援基盤プラットフォーム(BINDS)「無細胞タンパク質合成法、非天然型アミノ酸導入法等による高難度タンパク質の生産と解析(研究代表者:横山茂之)」、日本学術振興会(JSPS)科学研究費助成事業基盤研究C「非天然型リジン誘導体をプローブとしたケミカルバイオロジー(研究代表者:柳沢達男)」による支援を受けて行われました。
背景
地球上のあらゆる生物は、DNAの持つ遺伝情報に基づいて細胞内でタンパク質を合成しています。生物を形作り、生命活動を制御するタンパク質はわずか20種類のアミノ酸から構成されています。タンパク質のアミノ酸の並び方は、DNAの中のA、G、C、Tの4種類の塩基の並び方に対応しています。三つの塩基が一つのアミノ酸に対応しており、この3塩基の並びを「コドン」と呼びます。コドンが正しくタンパク質のアミノ酸配列に変換されるために、遺伝暗号の翻訳[4]が転移RNA(tRNA)[4]を介して行われます。
DNAの情報が伝令RNA(mRNA)に写し取られた後、その情報が正しくアミノ酸に翻訳されるには、特定のアミノ酸が特定のtRNAと結びつく必要があります。その両者を選び出して遺伝暗号表の通りに結合させるのが、「アミノアシルtRNA合成酵素(aaRS)[5]」と呼ばれる酵素群です。aaRSを遺伝子工学的に改変すると、遺伝暗号表を書き換えることができます。天然アミノ酸以外の新しいアミノ酸と新しいtRNAを結びつけるaaRSを作れば、非天然型アミノ酸(人工アミノ酸)を特定のコドンに結びつけることが可能です。
横山特別招聘研究員と坂本チームリーダーらはこれまでに、天然アミノ酸にはない機能を持つ人工アミノ酸をタンパク質に導入する技術[6]開発を進めてきました注1-5)。しかし既存のaaRSを改変して、宿主のaaRSと競合しない人工アミノ酸の導入システムを作り出すには、手間と時間がかかります。
一方で22番目のアミノ酸、ピロリシン[7]とそれを認識する「ピロリシルtRNA合成酵素(PylRS)」の発見により、あらゆる生物でさまざまな形の人工アミノ酸をタンパク質内に自在に導入することが可能になりました。共同研究グループは、ピロリシンの発見当初から、PylRSのX線結晶構造解析を進めており、2008年にPylRSのY306A/Y384F変異体「AF変異PylRS」を開発しました注1)。AF変異PylRSは、特に大きなサイズの人工アミノ酸を認識できることから、現在では世界中でタンパク質への導入に汎用され、大きな成果が得られています。
しかし、AF変異PylRSがどのように基質である人工アミノ酸を認識してtRNAに結合させるのかについてはこれまで詳細が分かっておらず、新しい人工アミノ酸の合理的な設計が困難でした。そのため、AF変異PylRSと人工アミノ酸との複合体の立体構造を、網羅的かつ系統的に決定することが求められていました。通常、多種類の基質を認識する酵素は、それらの共通部分を認識すると考えられます。しかしAF変異PylRSの基質になりうる人工アミノ酸は多様な形をしており、このような常識では説明できませんでした。
そこで、共同研究グループは,実際にAF変異PylRSとさまざまな形の基質アミノ酸との結合の様子を、原子レベルの分解能で網羅的に明らかにすることを試みました。
- 注1)
- Yanagisawa T et. al. Multistep engineering of pyrrolysyl-tRNA synthetase to genetically encode N(epsilon)-(o-azidobenzyloxycarbonyl) lysine for site-specific protein modification. Chem.Biol.15, 1187-1197 (2008).
- 注2)
- Sakamoto K et. al. Site-specific incorporation of an unnatural amino acid into proteins in mammalian cells. Nucleic Acids Res.30,4692-4699 (2002).
- 注3)
- Kiga D et. al. An engineered Escherichia coli tyrosyl-tRNA synthetase for site-specific incorporation of an unnatural amino acid into proteins in eukaryotic translation and its application in a wheat germ cell-free system. Proc.Natl.Acad.Sci U.S.A. 99,9715-9720 (2002).
- 注4)
- Mukai T et. al. Codon reassignment in the Escherichia coli genetic code. Nucleic Acids Res. 38,8188-8195 (2010).
- 注5)
- Kato A et. al. Extensive survey of antibody invariant positions for efficient chemical conjugation using expanded genetic codes. Bioconjug.Chem. 28,2099-2108 (2017).
研究手法と成果
共同研究グループは、AF変異PylRSと14種類の人工アミノ酸との複合体のX線結晶構造解析を、大型放射光施設「SPring-8」[8]の共用ビームラインを利用して、0.14ナノメートル(nm、1nmは10億分の1メートル)という原子レベルの分解能で行いました(図1)。
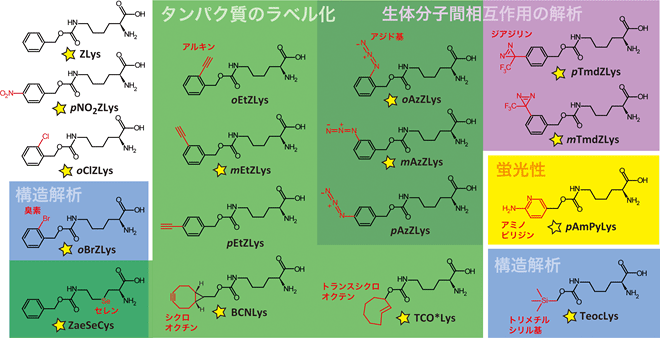
図1 本研究でタンパク質への導入を調べた17種類の人工アミノ酸
AF変異PylRSは、タンパク質のラベル化(アジド基、アルキンなど)、光クロスリンクによる生体分子間相互作用の解析(アジド基、ジアジリンなど)、構造解析(セレン、臭素、トリメチルシリル基)など、さまざまな用途に使える人工アミノ酸をタンパク質へ導入することができる。今回、17種類のうち14種類の結晶構造解析に成功した(黄色星印)。
その結果、AF変異PylRSは、活性部位の奥に大きな疎水性ポケットを持っており、主に疎水性の相互作用により、人工アミノ酸が疎水性ポケットに収納されていることが分かりました。このAF変異PylRSは、306番目のチロシン(Y)という大きなアミノ酸がアラニン(A)という小さなアミノ酸に置き換えられています。そのため、基質アミノ酸と結合するためのポケット(結合ポケット)が大きく広がっており、ベンゼン環を含むような大きな人工アミノ酸を受け入れることができます(図1、2)。
また、人工アミノ酸によっては、複数個結合した状態の組み合わせとしてAF変異PylRSに認識され、結合ポケットの隙間を埋める様子も観察できました(図2中・右)。さらに、アミノ酸の種類によって、結合ポケットの構造を変化させて対応しているものもありました(図2左)。つまり、AF変異PylRSがそれぞれの人工アミノ酸に特異的に対応し、かつ異なる様式でそれぞれを認識していることが分かりました。一般的に、20種類のaaRSは基質アミノ酸を厳密に識別するため、アミノ酸がaaRSの基質の鍵穴にはぴったりはまらず、結合ポケットに余ったスペースができると、そのアミノ酸とは結合できません。従って、通常はその人工アミノ酸にぴったりとはまる結合ポケットを持つaaRSが必要です。一方、AF変異PylRSは基質アミノ酸の形や大きさが多少異なっていても取り込むことができます。これは、その人工アミノ酸に最適な変異体を一つずつ作製しなくてもよいという、とても便利な特徴です。今回、AF変異PylRSの立体構造を解くことにより、その仕組みを明らかにすることができました。このように、AF変異PylRSが巧みにさまざまな人工アミノ酸を認識して結合する様子を網羅的、系統的に捉えたのは、世界でも類を見ない成果です。
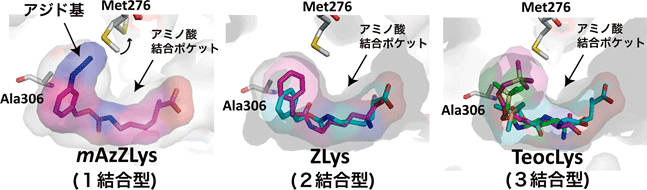
図2 AF変異PylRSと3種類の人工アミノ酸の複合体
AF変異PylRSとの複合体の構造解析に用いたもののうち、1結合型(mAzZLys:赤紫)、2結合型(ZLys:水色、赤紫)、3結合型(TeocLys:水色、緑、赤紫)の人工アミノ酸を示した。mAzZLysは、メタ位のアジド基が結合ポケットの隙間を埋めている。一方で、結合ポケットよりも少し小さいZLysやTeocLysは、結合ポケット内部でそれぞれが2個および3個結合した状態の組み合わせとして認識される。Met276(276番目のメチオニン残基)は人工アミノ酸の大きさに対応して向きを変え、ポケットの大きさを調節する。
今後の期待
今回得られた立体構造をもとにAF変異PylRSのアミノ酸結合ポケットを人工的にデザインすることで、今後はさらに広範囲な新しい人工アミノ酸を認識させられるようになると考えられます。こうして作られた変異PylRSには、特に医療の面での応用性を飛躍的に高めると考えられます。
例えば、コンピュータを用いた医薬品の分子設計により、従来以上の精度で新しい人工アミノ酸をtRNAに結合させられることで、あらゆる人工アミノ酸をタンパク質の材料として用いることができるようになります。こうして作られた高機能なタンパク質は、生命現象の解明や病気の診断、そして工業的に有用な酵素や体内で働く抗がんタンパク質の作製など、幅広い用途が期待できます。
論文情報
- タイトル
- Structural basis for genetic-code expansion with bulky lysine derivatives by an engineered pyrrolysyl-tRNA synthetase
- 著者名
- Tatsuo Yanagisawa*, Mitsuo Kuratani, Eiko Seki, Nobumasa Hino, Kensaku Sakamoto, Shigeyuki Yokoyama*
- 雑誌
- Cell Chemical Biology
- DOI
- 10.1016/j.chembiol.2019.03.008
補足説明
- [1]AF変異ピロリシルtRNA合成酵素(AF変異PylRS)
- ピロリシルtRNA合成酵素(PylRS)の結晶構造をもとに、研究グループが開発したY306A/Y384F変異型PylRS。AF変異PylRSは、基質特異性の厳しい一般的なアミノアシルtRNA合成酵素(aaRS)と異なり、様々な形の、特に大きなサイズの人工アミノ酸を取り込み、アミノアシル化することができる。
- [2]X線結晶構造解析
- 物質の結晶にX線を照射して回折データを解析することにより、物質の内部構造を調べる方法。タンパク質の構造を原子レベルの分解能で詳細に解明するための最も有力な方法の一つである。
- [3]基質
- 酵素によって化学反応を触媒される物質。酵素反応にとどまらず触媒反応全般に拡張して用いられたり、化学反応全般における原料物質という意味で使用されたりすることもある。
- [4]翻訳、トランスファーRNA(tRNA)
- 翻訳とは、DNAのA、G、C、チミン(T)の情報(塩基配列)をアミノ酸配列に変換しタンパク質を合成すること。伝令RNA(mRNA)の塩基配列であるアデニン(A)、グアニン(G)、シトシン(C)、ウラシル(U)は、翻訳の過程で3つが組(コドン)となって1つのアミノ酸を指定する。DNAの塩基配列の情報と、アミノ酸とを対応付けるアダプター分子がtRNAである。
- [5]アミノアシルtRNA合成酵素(aaRS)、アミノアシル化
- タンパク質を構成するアミノ酸は主に20種類存在する。20種類のアミノ酸のそれぞれに対してアミノアシルtRNA合成酵素が20種類存在し(アスパラギン酸に対応するAspRS、セリンに対応するSerRSなど)、ATPのエネルギーを利用して特定のアミノ酸を活性化したのち特定のtRNAのCCA末端に付加する(アミノアシル化)。
- aaRS + アミノ酸 + ATP + tRNA → aaRS・(アミノアシルAMP) + tRNA →アミノアシルtRNA + aaRS
- [6]非天然型アミノ酸(人工アミノ酸)の導入システム
- 人工アミノ酸は特定のaaRSによりtRNAに結合(アミノアシル化)したのち、リボソーム上で翻訳され、タンパク質内の特定の位置に部位特異的に導入される。通常はアミノ酸をコードしないUAGなどの終止コドンを利用して導入することが多い。チロシルtRNA合成酵素(TyrRS)とPylRSの系において最も開発が進んでおり、既に大腸菌、酵母、ショウジョウバエ、線虫、マウス、哺乳類細胞、シロイヌナズナなどを宿主として人工アミノ酸が導入されている。 詳しくは以下を参照。
- 「人工アミノ酸のタンパク質への部位特異的導入技術」(理化学研究所ライフサイエンス技術基盤研究センター)のHP
- 理研SSBC 拡張遺伝暗号システム研究チームのHP
- [7]ピロリシン
- 一部のメタン生成古細菌や細菌に存在し、UAGコドンによって規定されるアミノ酸の1種で、22番目のアミノ酸と呼ばれている。PylRSとtRNAPylの系により、メタン生成に必要なメチル基転移酵素の活性部位に導入され、活性に重要なアミノ酸残基として働く。構造はリシンと似ているが、側鎖の末端にピロリン環が付加している。
- [8]大型放射光施設「SPring-8」
- 理研が所有する、兵庫県の播磨科学公園都市にある第三世代放射光施設。放射光(シンクロトロン放射)とは、電子を光とほぼ等しい速度まで加速し、電磁石によって進行方向を曲げたときに発生する細くて強力な電磁波のこと。SPring-8では、遠赤外から可視光線、軟X線を経て硬X線に至る幅広い波長域で放射光を得ることができるため、原子核の研究からナノテクノロジー、バイオテクノロジー、産業利用や科学捜査まで幅広い研究が行われている。SPring-8はSuper Photon ring-8GeVの略。
発表者・機関窓口
発表者
理化学研究所 科技ハブ産連本部 バトンゾーン研究推進プログラム 横山特別研究室
特別招聘研究員 横山 茂之(よこやま しげゆき)
理化学研究所 生命機能科学研究センター 非天然型アミノ酸技術研究チーム
上級研究員(研究当時) 柳沢 達男(やなぎさわ たつお)
機関窓口
理化学研究所 広報室 報道担当
AMED事業
日本医療研究開発機構 創薬戦略部 医薬品研究課



