(HKU Biomedical Engineering develops novel 3D imaging technology to make fluorescence microscopy more efficient and push the boundaries of living cells research)
2020/4/9 香港大学 (HKU)
・ HKU が、新たな光学撮像技術、Coded Light-sheet Array Microscopy(CLAM)を開発。
・ エネルギー効率に優れ、生きた細胞等の被験物を走査中に優しく保護しながら高速 3D 蛍光撮像。
従来の蛍光顕微鏡では、生物活動の 3D 追跡には遅すぎること、また、強力な光の照射で生きた被験物を損傷するという問題があった。
・ 従来の 3D 蛍光顕微鏡プラットフォームでは、点、線、平面毎に被験物全体を連続走査・撮像する必要があるため動作が遅くなり、太陽光の数千~数百万倍強力な光の照射により被験物を損傷するため、解剖学や発生生物学、神経科学等、様々なアプリケーションでの長期的な生物撮像に適さない。
・ さらに、同プラットフォームでは、「フォトブリーチング(蛍光退色)」が被験物の撮像量を制限する問題もある。繰り返す光の照射で蛍光退色が進み、余剰に発生する蛍光は撮像の完成に使われず無駄になる。
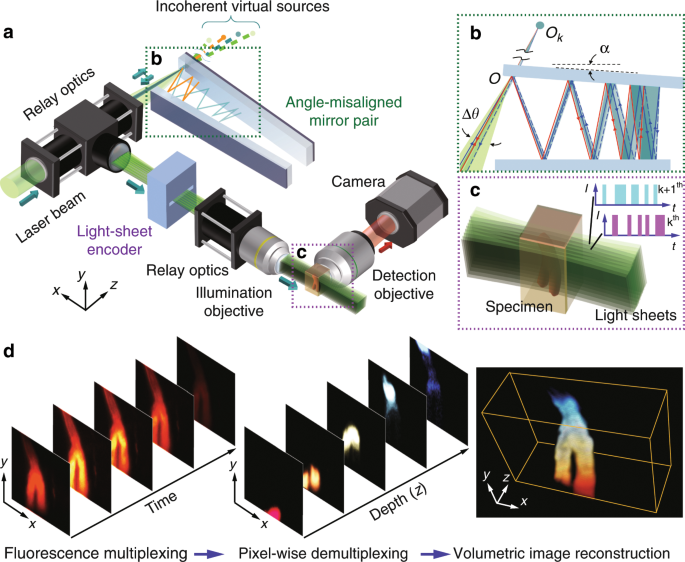
・ 新技術の CLAM では、一対のパラレルミラーにより一本のレーザービームを「光のシート」の高密度アレイに転換し、蛍光励起として被験物の大部分を覆う。これにより、従来のような点線平面毎の走査の必要が無く、3D 構造全体の画像を一度に捉えられる。また、一般的な 3D 蛍光撮像法に比して、フォトブリーチングの低減にも優れる。
・ 電気通信で複数の信号を同時に送信する際に利用される画像エンコード技術の符号分割多重化(Code Division Multiplexing: CDM)を活用し、CLAM の画像解像度と品質を確保。CDM により、2D 撮像センサーで全画像のスタックを一度に捕獲してデジタル的に 3D に再構築できるようになる。3D 撮像での同技術の利用は今回が初めて。
・ CLAM の概念実証として、マイクロ流体チップで高速移動するマイクロ粒子(10v/s 以上)の 3D 動画を撮像。CLAM には撮像速度の制限は無いが、スナップショットを撮影するカメラ等のシステム中の検出器の速度による制限がある。CLAM では、高速カメラ技術の進展に合わせ、より高速の走査達成を目指すことができる。
・ HKU の LKS 医学部が新たに開発した細胞透明化技術と CLAM を組合せ、マウスの腎糸球体と腸血管構造の高フレームレートでの 3D 可視化を実証。このような組み合わせにより、神経科学研究での脳の細胞組織のマッピングのような、記録保存用の生体サンプルの大規模な 3D 病理組織学的調査での利用も可能になる。
・ また、生きている被験物を損傷せず継続的に観察できることから、動物の胚の成長過程の追跡や、細胞・有機体の細菌やウィルスによる感染のリアルタイムモニタリング等、生物細胞学のあらゆる側面での基本的な理解に影響を及ぼす可能性も考えられる。
・ CLAM は、最少のハードウェアとソフトウェアであらゆる顕微鏡システムに取り入れることが可能。生物細胞学や動植物発生生物学の研究に向け、現行のシステムをアップグレードする予定。
・ 本技術は米国特許出願済み。本研究には、HKSAR Research Grants Council、Innovation and Technology Support Program、HKU の University Development Funds および中国国家自然科学基金(NSFC)が資金を提供した。
URL: https://www.hku.hk/press/press-releases/detail/20876.html
<NEDO海外技術情報より>
(関連情報)
Light: Science & Applications 掲載論文(フルテキスト)
Parallelized volumetric fluorescence microscopy with a reconfigurable coded incoherent light-sheet
array
URL: https://www.nature.com/articles/s41377-020-0245-8
Abstract
Parallelized fluorescence imaging has been a long-standing pursuit that can address the unmet need for a comprehensive three-dimensional (3D) visualization of dynamical biological processes with minimal photodamage. However, the available approaches are limited to incomplete parallelization in only two dimensions or sparse sampling in three dimensions. We hereby develop a novel fluorescence imaging approach, called coded light-sheet array microscopy (CLAM), which allows complete parallelized 3D imaging without mechanical scanning. Harnessing the concept of an “infinity mirror”, CLAM generates a light-sheet array with controllable sheet density and degree of coherence. Thus, CLAM circumvents the common complications of multiple coherent light-sheet generation in terms of dedicated wavefront engineering and mechanical dithering/scanning. Moreover, the encoding of multiplexed optical sections in CLAM allows the synchronous capture of all sectioned images within the imaged volume. We demonstrate the utility of CLAM in different imaging scenarios, including a light-scattering medium, an optically cleared tissue, and microparticles in fluidic flow. CLAM can maximize the signal-to-noise ratio and the spatial duty cycle, and also provides a further reduction in photobleaching compared to the major scanning-based 3D imaging systems. The flexible implementation of CLAM regarding both hardware and software ensures compatibility with any light-sheet imaging modality and could thus be instrumental in a multitude of areas in biological research.


_no%20white%20border_30f0231a84.jpg)