2023-02-22 アメリカ合衆国・ワシントン大学(UW)
学習(ML)アルゴリズムを開発。医療から工業生産まで広く利用可能な酵素のため、タンパク質の設計技術における重要な進展となる。
・ ML ソフトウェアを利用し、ルシフェリンと反応する数千種類のタンパク質構造を生成。研究室での試験において効率的な発光酵素を特定し、「Lux Sit(ラテン語で「光あれ」の意)」と命名。
・ 最適化された同発光酵素では性能が飛躍的に向上。ウミシイタケの天然のルシフェラーゼよりも明るく発光し、裸眼でも確認できる。この技術ブレイクスルーは、原則的にほとんどの化学反応に向けたカスタム酵素の設計が可能であることを示している。
・ 新しい酵素は、例えばバイオテクノロジー分野ではバイオ燃料生産、食品加工や医薬品製造の向上に、また、医療分野では治療・診断ツールとして役立てられる。さらに、汚染物質の分解や汚染エリアの浄化による環境の修復や、生分解性プラスチックや接着剤等の新しい材料の開発も支援する。
・ 本研究は、ハワード・ヒューズ医学研究所(HHMI)、米国立衛生研究所(NIH)、 United World Antiviral
Research Network、米国国立アレルギー・感染症研究所(NIAID)、Institute for Protein Design の
Audacious Project、Open Philanthropy Project Improving Protein Design Fund、デンマーク Novo Nordisk Foundation、米国立科学財団(NSF)およびシュミッツ・フューチャーズプログラムが支援した。中国国家自然科学金委員会がコンピューティングリソースを一部提供。
URL: https://newsroom.uw.edu/news/ai-conjures-proteins-speed-chemical-reactions
<NEDO海外技術情報より>
関連情報
Nature 掲載論文(フルテキスト)
De novo design of luciferases using deep learning
URL: https://www.nature.com/articles/s41586-023-05696-3
Abstract
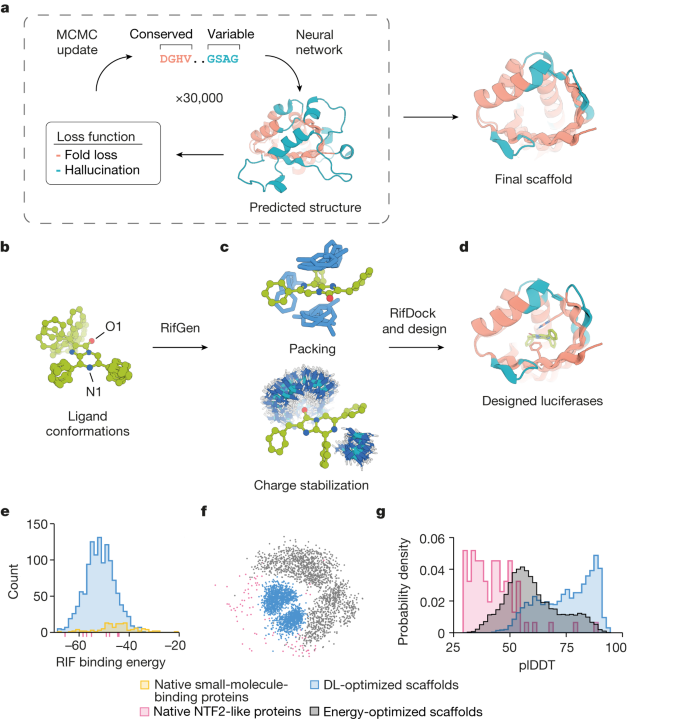
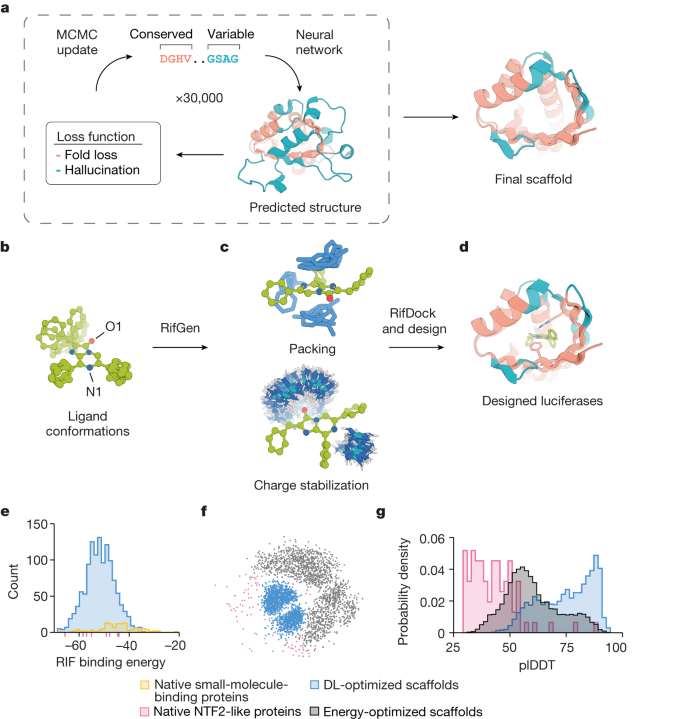
De novo enzyme design has sought to introduce active sites and substrate-binding pockets that are predicted to catalyse a reaction of interest into geometrically compatible native scaffolds1,2, but has been limited by a lack of suitable protein structures and the complexity of native protein sequence–structure relationships. Here we describe a deep-learning-based ‘family-wide hallucination’ approach that generates large numbers of idealized protein structures containing diverse pocket shapes and designed sequences that encode them. We use these scaffolds to design artificial luciferases that selectively catalyse the oxidative chemiluminescence of the synthetic luciferin substrates diphenylterazine3 and 2-deoxycoelenterazine. The designed active sites position an arginine guanidinium group adjacent to an anion that develops during the reaction in a binding pocket with high shape complementarity. For both luciferin substrates, we obtain designed luciferases with high selectivity; the most active of these is a small (13.9 kDa) and thermostable (with a melting temperature higher than 95 °C) enzyme that has a catalytic efficiency on diphenylterazine (kcat/Km = 106 M−1 s−1) comparable to that of native luciferases, but a much higher substrate specificity. The creation of highly active and specific biocatalysts from scratch with broad applications in biomedicine is a key milestone for computational enzyme design, and our approach should enable generation of a wide range of luciferases and other enzymes.