2025-08-07 中国科学院(CAS)
中国科学院遺伝発育生物学研究所の馮健博士と韓方普博士らは、マメ科モデル植物Medicago truncatula(A17)とM. littoralis(R108)の初のギャップなしテロメア間(T2T)全ゲノムを構築し、植物セントロメアの進化を解明した(Molecular Plant誌)。PacBio HiFi、Oxford Nanopore超長鎖リード、Hi-C解析により、A17は494.47Mb、R108は415.27Mbで完全性>99%を達成。セントロメア解析ではA17がCentM168と種特異的CentM183で構成され、R108はほぼCentM168のみと判明。CENH3富化CentM168配列が活性を維持し、衛星DNA豊富な周辺領域が安定性を高める可能性を示した。若いLTRレトロトランスポゾンの集積も確認され、本成果はマメ科ゲノム研究や精密育種の基盤となる。
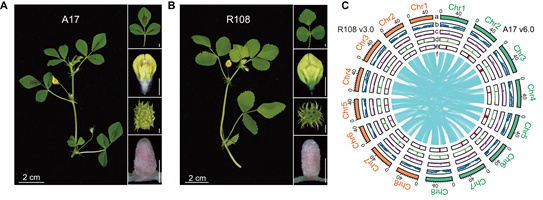
Phenotypicvariations observed between A17 and R108 (Image by IGDB)
<関連情報>
- https://english.cas.cn/newsroom/research_news/life/202508/t20250807_1049279.shtml
- https://www.sciencedirect.com/science/article/pii/S167420522500245X
2つの完全なテロメアからテロメアまでのMedicagoゲノムが、セントロメアの構造と進化の全体像を明らかにする Two complete telomere-to-telomere Medicago genomes reveal the landscape and evolution of centromeres
Lisha Shen, Congyang Yi, Yang Liu, Fangpu Han, Jian Feng
Molecular Plant Available online: 25 July 2025
DOI:https://doi.org/10.1016/j.molp.2025.07.016
Short Summary
In this study, we report two gap-free genome assemblies of Medicago truncatula A17 and Medicago littoralis R108 generated using long-read sequencing. The assemblies reveal distinct centromere compositions, highlighting lineage-specific satellite repeat evolution and providing valuable resources for legume functional genomics and centromere biology.