2024-06-20 アイルランド・ダブリン大学トリニティ・カレッジ
- ダブリン大学トリニティ・カレッジ、マサチューセッツ工科大学(MIT)・ハーバード大学ブロード研究所およびハーバード大学医学大学院が、β-ラクタマーゼの合理的な遺伝子操作を可能にするアルゴリズムを開発。
- 酵素は生命にとって不可欠なものであり、社会の課題に対処する革新的な医薬品やツールの開発の鍵となるもの。その立体構造を支える数百個のアミノ酸配列の変化を通じ、数十億年をかけて進化を遂げている。
- 酵素は立体構造を形成することで、食物タンパク質の消化や化学エネルギーの筋肉の力への変換等の特定の機能を担う。アミノ酸の配列が変更されると立体構造が崩壊して酵素の機能性が変化し、完全に無効になることもある。
- 酵素の活性を向上させるために、分子生物学の最新のツールを用いてアミノ酸配列に変更を加えることは簡単でコスト効率が高いが、僅か 3~4 個のランダムな配列変更で酵素の活性が大きく失われる可能性がある。
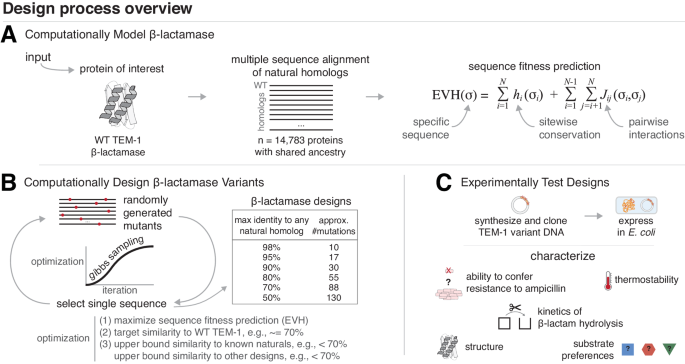
- 本研究では、ランダムな変異を導入する方法に代わり、酵素の進化の歴史を考慮したアルゴリズムによる新手法を開発。同アルゴリズムは様々な生物のβ-ラクタマーゼの数千種類もの配列を利用するスコアリング関数で、280 の配列で最大 84 の突然変異を生成して機能性能を向上させるもの。
- 新手法で設計された酵素では、より高温での活性と安定性が向上。X 線結晶構造解析でその立体構造を確認すると、アミノ酸の 30%が変化しているにもかかわらず、野生型β-ラクタマーゼと同一の構造であることがわかった。
- また、通常は個々のアミノ酸の変化が酵素構造を損なわせることとは対照的に、同時に取り入れられたアミノ酸の調整された変化が立体構造を効率的に安定させることもわかった。
- 酵素を必要とする食品製造プロセス、プラスチック分解酵素や人間の健康や疾病に関連するプロセス等、産業界での幅広いアプリケーションの可能性が期待できる。
- 本研究は、米国立衛生研究所(NIH)の一般医科学研究所(NIGMS)、アイルランド科学財団(SFI)、米国エネルギー省(DOE) Biological and Environmental Research Program、ハーバード大学医学大学院 SynBio HIVE およびダナ・ファーバー癌研究所が支援した。
URL: https://www.tcd.ie/news_events/articles/2024/scientists-devise-algorithm-to-engineer-improved-enzymes/
<NEDO海外技術情報より>
関連情報
Nature Communications 掲載論文(フルテキスト)
Simultaneous enhancement of multiple functional properties using evolution-informed protein design
URL: https://www.nature.com/articles/s41467-024-49119-x
Abstract
A major challenge in protein design is to augment existing functional proteins with multiple property enhancements. Altering several properties likely necessitates numerous primary sequence changes, and novel methods are needed to accurately predict combinations of mutations that maintain or enhance function. Models of sequence co-variation (e.g., EVcouplings), which leverage extensive information about various protein properties and activities from homologous protein sequences, have proven effective for many applications including structure determination and mutation effect prediction. We apply EVcouplings to computationally design variants of the model protein TEM-1 β-lactamase. Nearly all the 14 experimentally characterized designs were functional, including one with 84 mutations from the nearest natural homolog. The designs also had large increases in thermostability, increased activity on multiple substrates, and nearly identical structure to the wild type enzyme. This study highlights the efficacy of evolutionary models in guiding large sequence alterations to generate functional diversity for protein design applications.