2018/06/20 国立遺伝学研究所
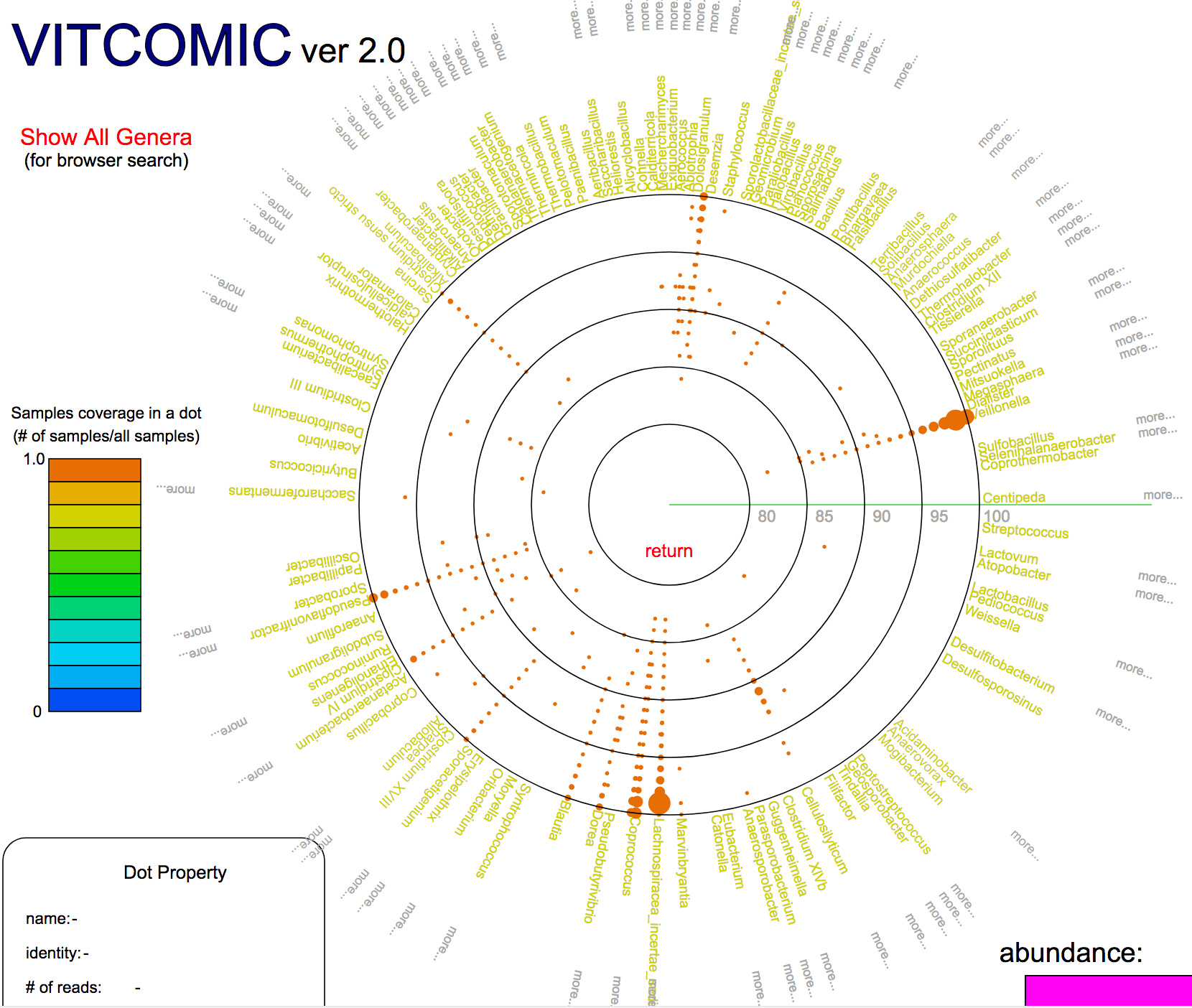
VITCOMIC2: visualization tool for the phylogenetic composition of microbial communities based on 16S rRNA gene amplicons and metagenomic shotgun sequencing.
Mori H, Maruyama T, Yano M, Yamada T, Kurokawa K. BMC Syst Biol
BMC Systems Biology 12 (Suppl 2), 30, 2018. DOI:10.1186/s12918-018-0545-2
情報・システム研究機構国立遺伝学研究所 生命情報研究センター ゲノム進化研究室 森宙史助教および黒川顕教授らは、細菌群集由来のDNAを丸ごとシーケンスすることで、個々の細菌を培養することなく解析可能な手法であるメタゲノム解析、および16S rRNA遺伝子のみをPCR増幅してシーケンスするメタ16S解析により得られる膨大なDNA配列データから、高速かつ高精度に細菌群集の系統組成を推定するウェブアプリケーション、VITCOMIC2を開発し公開しました。VITCOMIC2では、DNA配列間の配列類似性をGraphics Processing Unit (GPU)を用いて高速に検索するツールCLASTを用いることで、各配列の高速な系統推定を実現しています。また、VITCOMIC2ではメタ16S解析等で一般的に用いられている配列クラスタリングを行わずに、全配列を配列類似性検索することによって、配列をクラスタリングで生じる系統推定結果の曖昧性を回避でき、高精度な系統推定を実現しています。VITCOMIC2は、http://vitcomic.org/から利用できます。
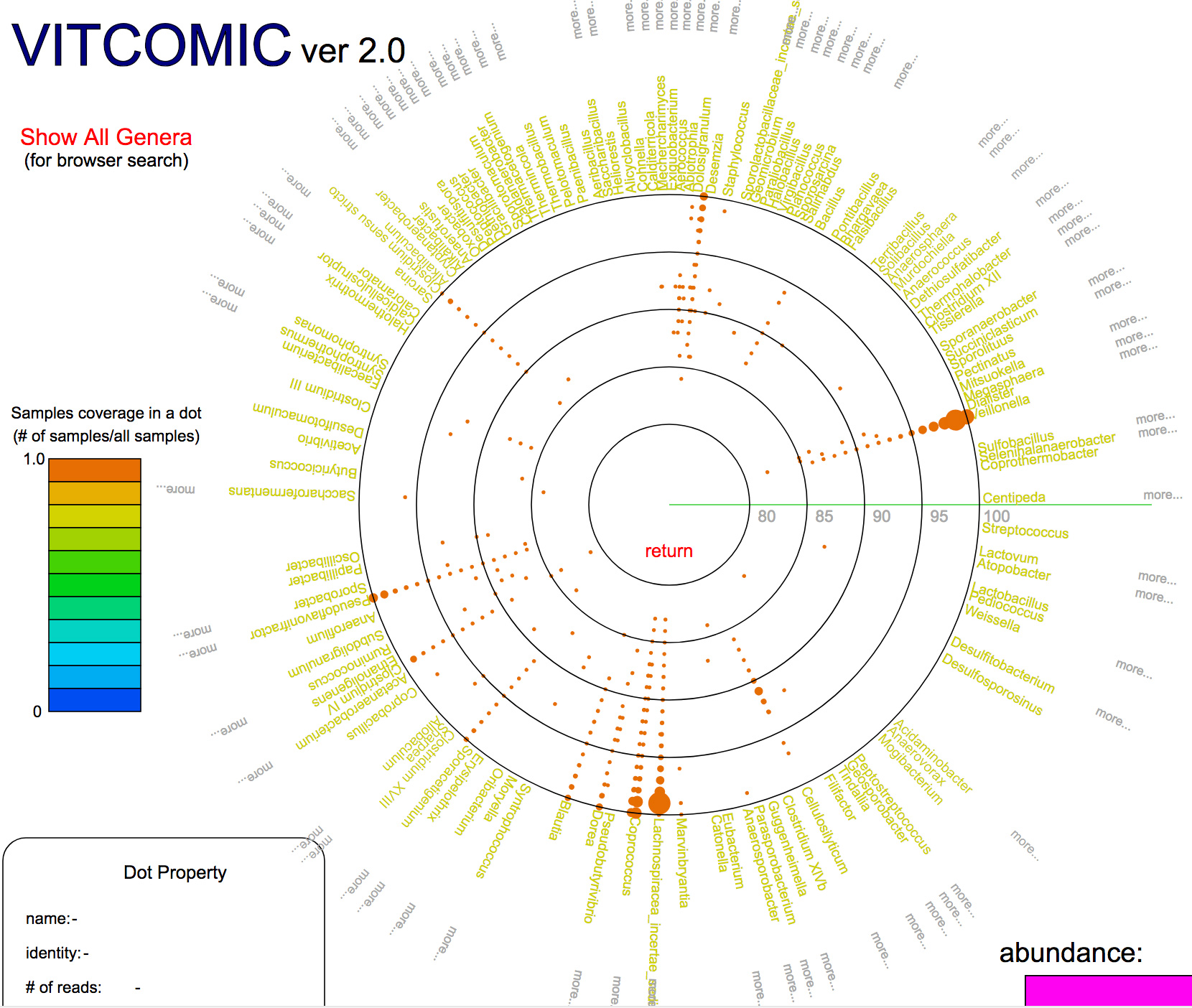
図:VITCOMIC2の結果の表示の例